|
Tabela 1. Dados clínicos dos pacientes |
||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||
|
Legenda: TCRBOA = Texas Cancer Research Biobank Open Access. Nota: A coluna celularidade tumoral na amostra indica o percentual do tecido da amostra que corresponde ao tumor. No paciente TCRBOA5, apenas 5% do tecido é tecido tumoral. |
Os tumores estavam localizados na cabeça do pâncreas, possuíam estágio T3, o que indicou que o tumor cresceu para além da glândula pancreática, N1, com acometimento de linfonodos, M0, e sem metástases, com exceção do paciente TCRBOA5 em quem não pôde ser determinada a presença ou ausência de metástase.
A qualidade do WES das amostras normal e tumoral é adequada, conforme análise do software FastQC27: todas com mais de 51 milhões de sequências de 101 bases, com conteúdo CG entre 45% e 46% e sem bases de baixa qualidade. Como consequência, o BWA26 realizou um bom alinhamento ao genoma de referência, GRCh38, conforme mostra a Tabela 2.
|
Tabela 2. Qualidade do alinhamento por amostra |
|||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||
|
Fonte: Log de processamento do software BWA26. Legenda: TCRBOA = Texas Cancer Research Biobank Open Access. Nota: O software BWA alinha os reads de 100 pares de bases ao genoma de referência GRCh38. Para cada amostra, são informados a quantidade de reads de 100 pares de base produzidos pelos sequenciadores Illumina (coluna quantidade total de reads), quantos desses reads o BWA conseguiu alinhar ao genoma de referência (coluna quantidade de reads alinhados), quantos ele não conseguiu alinhar (coluna quantidade de reads não alinhados) e o percentual de reads alinhados. Os índices de alinhamento são superiores a 99% para todas as amostras, o que indica uma boa qualidade. A denominação da amostra é composta pela identificação do paciente seguida da terminação T ou N, conforme seja a amostra de tecido tumoral, ou normal, respectivamente. Desse modo, TCRBOA1-T identifica a amostra tumoral do paciente TCRBOA1. |
Após o alinhamento, na etapa de identificação de variantes, os softwares GATK e Mutec2 encontraram 123.174 variantes, sendo 87.170 germinativas e 36.004 somáticas. Os dados por paciente, amostra e tipo de tecido estão na Tabela 3.
|
Tabela 3. Quantificação das variantes por amostra e tipo |
|||||||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||||||
|
Legendas: TCRBOA = Texas Cancer Research Biobank Open Access; SNP = base nitrogenada do genoma; INDEL = pequenas inserções ou deleções de algumas bases nitrogenadas no genoma. Nota: Cada paciente teve duas amostras tumorais sequenciadas: uma do tecido tumoral (PDAC) e outra de tecido saudável (glóbulos brancos). Uma vez alinhadas ao genoma de referência GRCh38, o software GATK identificou, para cada uma delas, as variantes presentes nas amostras. A tabela quantifica para cada amostra as variantes do tipo SNP, no qual apenas uma base está alterada no genoma da amostra, e do tipo INDEL, no qual diversas bases foram nele removidas ou inseridas. |
|||||||||||||||||||||||||||||||||||||||||||||||||
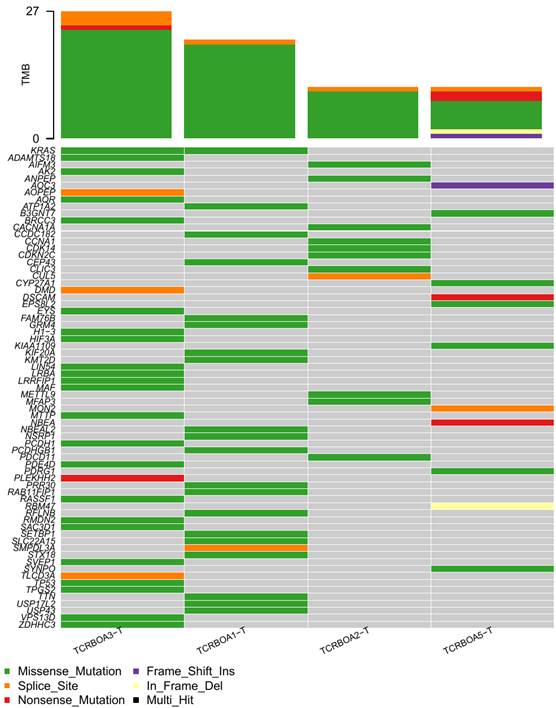
Entre as variantes SNP e INDEL somáticas encontradas, 69 são de alta qualidade e estão mostradas na Figura 1, agrupadas por paciente. O gene KRAS está mutado em dois dos quatro pacientes. Mutações do tipo missense, nas quais há alteração na proteína capaz ou não de afetar sua função, e tipo nonsense, nas quais a alteração da proteína causa uma provável perda de função, são as mais prevalentes. Ademais, entre as variantes somáticas, três são de alto impacto (causam perda da função da proteína: genes AOPEP, DMD e DSCAM) e cinco patogênicas (genes KRAS, TP53, DMD e CYP27A1)35. A Figura 1 ilustra as variantes somáticas do tipo SNP e INDEL agrupadas por tumor, o que mostra de forma didática a diversidade e especificidade tumoral em nível molecular.
|
|
|
Figura 1. Variantes somáticas encontradas nas amostras de PDAC dos pacientes Nota: A coluna à esquerda indica o nome do gene que apresenta mutações. Na linha, a identificação do paciente. A cor identifica o tipo de mutação: mutações do tipo missense, nas quais a mudança na base muda o códon do aminoácido, o que pode ou não prejudicar a função da proteína, e são as mais prevalentes. Nas mutações nonsense, a função da proteína é prejudicada pela mutação. A amostra TCRBOA5-T possui baixa celularidade tumoral, o que prejudica a análise. |
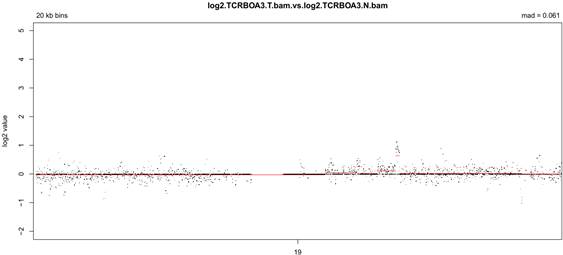
Das variantes estruturais (SV), identificou-se uma duplicação cromossômica na região 19q13.2, com aproximadamente 418 mil bases (Figura 2). Essa duplicação atingiu os genes MAP3K10, CNTD2, AKT2, C19orf47, PLD3, HIPK4, PRX, SERTAD1, BLVRB, SPTBN436.
|
|
|
Figura 2. Alterações estruturais presentes no cromossomo 19 na amostra tumoral do paciente TCRBOA3 Nota: A região duplicada aparece como uma linha paralela horizontal. Essa região corresponde ao segmento 19q13.2 do cromossomo 19 e tem aproximadamente 418 mil bases. Entre os genes contidos nessa região está o AKT2, oncogene com evidência na literatura de associação com maior instabilidade cromossômica do tumor. |
Das variantes germinativas, tem-se 14 variantes patogênicas, sendo uma associada à pancreatite crônica (gene CFTR, variante rs113993960, deleção, paciente TCRBOA3); 93 variantes associadas a fatores de risco para doenças como: síndrome de Von-Hippel-Lindau (gene CCND1, variante rs9344, genótipo A/A, paciente TCRBOA2); e pancreatite hereditária (gene CTRC, variante rs121909239, genótipo C/T, paciente TCRBOA3)35. Por fim, as variantes associadas à melhor resposta a medicamentos: para os compostos de platina, o paciente TCRBOA5 apresenta as variantes rs1042522, gene TP53, genótipo C/C (polimorfismo do códon 72) e variante rs25487, gene XRCC1, genótipo C/C35.
A partir desses resultados, torna-se factível avaliar as possibilidades de terapia-alvo para os quatro pacientes, conforme as características moleculares das células contidas nas amostras tumorais e normais.
Discussão
Conforme a Tabela 1, os dados clínicos dos pacientes sugerem tumores semelhantes e, embora não haja informações sobre a aptidão dos pacientes para a quimioterapia, o protocolo estabelece abordagens análogas de terapêutica: cirurgia mais quimioterapia neoadjuvante e adjuvante com FOLFIRINOX6. Ocorre que, conforme os resultados obtidos a partir do WES, observa-se que, apesar de histologicamente similares, em nível molecular, os tumores dos quatro pacientes em análise são bem diversos, com importantes implicações na escolha do tratamento.
O PDAC do paciente TCRBOA3 apresenta duplicação do segmento 19q13.2, onde se localiza o gene AKT2. Esse oncogene pertence à via de sinalização do gene PI3K, associada ao crescimento, proliferação, sobrevivência e invasão celular, bem como resistência à quimioterapia e prognóstico reservado. Desse modo, é indicado avaliar o uso de terapias-alvo que inibam a via PI3K/AKT/mTOR37. Ainda, esse paciente possui mutação do gene KRAS (variante rs121913529, genótipo C/T) que também promove proliferação e sobrevivência celular. Logo, terapias-alvo de inibição da via RAS38 também estariam indicadas.
Em uma outra abordagem terapêutica, a presença da duplicação do segmento 19q13.2, somada ao elevado número de mutações somáticas, sugere tratar-se de um tumor com instabilidade estrutural, cuja resposta aos inibidores de PARP associados a quimioterápicos com platina é boa3,18. Ademais, esse paciente possui dois polimorfismos associados à pancreatite crônica (genes CFTR, variante rs113993960 e CTRC, variante rs121909239)35, o que indica o rastreio do PDAC nos seus ascendentes e descendentes.
Um quadro diverso se desenha para o paciente TCRBOA2. No seu tumor, a análise das variantes somáticas revela alterações nos genes associados à via das ciclinas/quinases dependentes de ciclinas (CDK): CCNA1, CDK14 e CDKN2C, que controlam a progressão do ciclo celular e cujas mutações resultam em proliferação celular desregulada e na formação de células-tronco, ambos mecanismos associados a um processo oncogênico. Assim, há a possibilidade do uso de terapias-alvo que inibam a via das CDK39 para impedir o crescimento tumoral. Esse fato, associado à pouca quantidade de mutações, e a ausência de alterações genômicas estruturais relevantes sugerem estabilidade genômica desse tumor, desaconselhando assim o uso de inibidores de PARP3,18. Não menos importante é a variante germinativa associada à síndrome de Von-Hippel-Lindau (gene CCND1 variante rs9344)35, o que também indica o rastreio do PDAC para os ascendentes e descendentes desse paciente.
Por sua vez, as variantes somáticas encontradas no tumor do TCRBOA5 não apresentaram nenhuma associação com aquelas documentadas como indutoras de PDAC. A razão disso pode ser a baixa celularidade tumoral presente na amostra sequenciada, de apenas 5%. Nessa análise, foram encontradas mutações do tipo nonsense nos genes NBEA e DSCAM, uma deleção no gene RBM47, genes KRAS e TP53 selvagens e ausência de alterações estruturais relevantes. Em relação às germinativas, esse paciente é portador de variantes associadas a uma melhor resposta a compostos de platina e melhor sobrevida (gene TP53, variante rs1042522, C/C e gene XRCC1, variante rs25487, C/C)35.
Por fim, o paciente TCRBOA1 apresentou mutações somáticas do tipo missense, entre elas a variante rs121913529, gene KRAS, genótipo T, cujo significado é patogênico e associado ao PDAC35. KRAS é um oncogene e pacientes com mutações apresentam mau prognóstico e baixa resposta à quimioterapia. De modo análogo ao paciente TCRBOA3, terapias-alvo inibidoras de KRAS são indicadas para o tratamento desse tumor. Uma outra característica é a ausência de instabilidade estrutural no genoma, o que contraindica os inibidores de PARP ou a imunoterapia3,18.
Um outro aspecto a ser analisado é que, a partir dos elementos vistos anteriormente, foi possível identificar características moleculares do tumor e do paciente que afetam o valor preditivo positivo do regime quimioterápico padrão. No paciente TCRBOA3, cujo AKT2 está duplicado, o uso do FOLFIRINOX sem inibir esse gene seria pouco eficaz; raciocínio análogo aplica-se ao paciente TCRBOA2, cuja via das CDK está permanentemente ativada.
Desse modo, para diminuir a morbimortalidade do PDAC, é necessário conhecê-lo em nível molecular, a fim de conformar a intervenção às características do tumor e do paciente. Para tanto, o WES mostra-se uma ferramenta adequada para alcançar esse objetivo.
Conclusão
O presente artigo apresentou todos os passos e as ferramentas necessários à execução da análise do exoma para identificar as variantes somáticas, germinativas e alterações estruturais no genoma do PDAC. A metodologia aqui descrita pode ser usada em quaisquer outras neoplasias e, embora realizada in silico, não só chegou aos mesmos oncogenes indutores de tumores anteriormente identificados nos pacientes TCRBOA1, TCRBOA2, TCRBOA3 e TCRBOA5, como também identificou uma inédita duplicação na região 19q13.2, onde se localiza o oncogene AKT2, no tumor do paciente TCRBOA3.
Também mostrou que, apesar de o estadiamento classificar uniformemente todos os PDAC, cada paciente – bem como o respectivo tumor – apresenta especificidades moleculares que afetam o diagnóstico e o desfecho clínico. A técnica de WES permite identificar essas especificidades e ampliar as possibilidades terapêuticas a partir do conhecimento das variantes somáticas, germinativas e alterações estruturais do genoma dos tumores. E que, considerando o preço do WES (aproximadamente 300 dólares), a relação custo-benefício é adequada, pois permite identificar as variantes somáticas e germinativas em um único exame. É necessário, contudo, reconhecer que essa técnica exige a formação de laboratórios específicos e com pessoal altamente treinado, incluindo bioinformática.
Por fim, pelas razões já expostas, fica clara a necessidade de divulgar e ampliar o uso da análise do WES nos serviços oncológicos, bem como capacitar as equipes na sua realização e utilização.
Ambos os autores contribuíram substancialmente na concepção e/ou no planejamento do estudo; na obtenção, análise e interpretação dos dados; na redação e revisão crítica; e aprovaram a versão final a ser publicada.
DECLARAÇÃO DE CONFLITO DE INTERESSES
Nada a declarar.
FONTES DE FINANCIAMENTO
Não há.
REFERÊNCIAS
1. Instituto Nacional de Câncer [Internet]. Rio de Janeiro: INCA; [data desconhecida]. Tipos de câncer: câncer de pâncreas; [atualizada 2021 ago 24; acesso 2022 jan 15]. Disponível em: https://www.inca.gov.br/tipos-de-cancer/cancer-de-pancreas
2. Siegel RL, Miller KD, Jemal A. Cancer statistics, 2020. CA Cancer J Clin. 2020;70(1):7-30. doi: https://doi.org/10.3322/caac.21590
3. Markham MJ, Wachter K, Agarwal N, et al. Clinical cancer advances 2020: annual report on progress against cancer from the American Society of Clinical Oncology. J Clin Oncol. 2020;38(10):1081. doi: https://doi.org/10.1200/JCO.19.03141. Erratum in: J Clin Oncol. 2020;38(26):3076. doi: https://doi.org/10.1200/JCO.20.02291
4. Luchini C, Capelli P, Scarpa A. Pancreatic ductal adenocarcinoma and its variants. Surg Pathol Clin. 2016;9(4):547-60. doi: https://doi.org/10.1016/j.path.2016.05.003
5. Porta M, Fabregat X, Malats N, et al. Exocrine pancreatic cancer: symptoms at presentation and their relation to tumour site and stage. Clin Transl Oncol. 2005;7(5):189-97. doi: https://doi.org/10.1007/BF02712816
6. National Comprehensive Cancer Network. Pancreatic adenocarcinoma [Internet]. Version 2.2021. Plymouth Meeting, PA: NCCN; 2021 Feb 25. [cited 2021 Oct 7]. Available from: https://www.nccn.org/guidelines/nccn-guidelines/guidelines-detail?category=1&id=1455
7. Amin MB, Edge S, Greene F, et al, editors. AJCC cancer staging manual. 8th ed. Springer International Publishing: American Joint Commission on Cancer; 2017.
8. Schmied BM, Z'graggen K, Redaelli CA, et al. Problems in staging of pancreatic and hepatobiliary tumours. Ann Oncol. 2000;11 Suppl 3:161-4. doi: https://doi.org/10.1093/annonc/11.suppl_3.161
9. Kamarajah SK, Burns WR, Frankel TL, et al. Validation of the American Joint Commission on Cancer (AJCC) 8th edition staging system for patients with pancreatic adenocarcinoma: a Surveillance, Epidemiology and End Results (SEER) Analysis. Ann Surg Oncol. 2017;24(7):2023-30. doi: https://doi.org/10.1245/s10434-017-5810-x
10. Cancer Genome Atlas Research Network, Weinstein JN, Collisson EA, et al. The Cancer Genome Atlas Pan-Cancer analysis project. Nat Genet. 2013;45(10):1113-20. doi: https://doi.org/10.1038/ng.2764
11. Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell. 2011;144(5):646-74. doi: https://doi.org/10.1016/j.cell.2011.02.013
12. McCombie WR, McPherson JD, Mardis ER. Next-generation sequencing technologies. Cold Spring Harb Perspect Med. 2019;9(11):a036798. doi: https://doi.org/10.1101/cshperspect.a036798
13. van Dijk EL, Auger H, Jaszczyszyn Y, et al. Ten years of next-generation sequencing technology. Trends Genet. 2014;30(9):418-26. doi: https://doi.org/10.1016/j.tig.2014.07.001
14. Ding L, Bailey MH, Porta-Pardo E, et al. Perspective on oncogenic processes at the end of the beginning of cancer genomics. Cell. 2018;173(2):305-20.e10. doi: https://doi.org/10.1016/j.cell.2018.03.033
15. Strom SP. Current practices and guidelines for clinical next-generation sequencing oncology testing. Cancer Biol Med. 2016;13(1):3-11. doi: https://doi.org/10.28092/j.issn.2095-3941.2016.0004
16. Cancer Genome Atlas Research Network. Integrated genomic characterization of pancreatic ductal adenocarcinoma. Cancer Cell. 2017;32(2):185-203.e13. doi: https://doi.org/10.1016/j.ccell.2017.07.007
17. Waddell N, Pajic M, Patch AM. et al. Whole genomes redefine the mutational landscape of pancreatic cancer. Nature. 2015;518(7540):495-501. doi: https://doi.org/10.1038/nature14169
18. Dreyer SB, Upstill-Goddard R, Paulus-Hock V, et al. Targeting DNA damage response and replication stress in pancreatic cancer. Gastroenterology. 2021;160(1):362-77.e13. doi: https://doi.org/10.1053/j.gastro.2020.09.043
19. Dreyer SB, Pinese M, Jamieson NB, et al. Precision oncology in surgery: patient selection for operable pancreatic cancer. Ann Surg. 2020;272(2):366-76. doi: https://doi.org/10.1097/SLA.0000000000003143
20. Ychou M, Conroy T, Seitz JF, et al. An open phase I study assessing the feasibility of the triple combination: oxaliplatin plus irinotecan plus leucovorin/ 5-fluorouracil every 2 weeks in patients with advanced solid tumors. Ann Oncol. 2003;14(3):481-9. doi: https://doi.org/10.1093/annonc/mdg119
21. Amstutz U, Henricks LM, Offer SM, et al. Clinical Pharmacogenetics Implementation Consortium (CPIC) guideline for dihydropyrimidine dehydrogenase genotype and fluoropyrimidine dosing: 2017 update. Clin Pharmacol Ther. 2018;103(2):210-6. doi: https://doi.org/10.1002/cpt.911
22. Swen JJ, Nijenhuis M, de Boer A, et al. Pharmacogenetics: from bench to byte--an update of guidelines. Clin Pharmacol Ther. 2011;89(5):662-73. doi: https://doi.org/10.1038/clpt.2011.34
23. Whirl-Carrillo M, McDonagh EM, Hebert JM, et al. Pharmacogenomics knowledge for personalized medicine. Clin Pharmacol Ther. 2012;92(4):414-7. doi: https://doi.org/10.1038/clpt.2012.96
24. PharmGKB [Internet]. Stanford (CA): Stanford University; c2001-2022. Clinical Annotation for UGT1A1*1, UGT1A1*28; FOLFIRI or irinotecan; Neutropenia (level 1A Toxicity); [cited 2022 Mar 13]. Available from: https://www.pharmgkb.org/clinicalAnnotation/1451204660
25. Becnel LB, Pereira S, Drummond JA, et al. An open access pilot freely sharing cancer genomic data from participants in Texas. Sci Data. 2016;3:160010. doi: https://doi.org/10.1038/sdata.2016.10
26. Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 2009;25(14):1754-60. doi: https://doi.org/10.1093/bioinformatics/btp324
27. FastQC [Internet]. Version 0.11.9. Cambridge: Babraham Institute. [2010]. – [cited 2022 Jan 3]. Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/
28. Benjamin D, Sato T, Cibulskis K, et al. Calling somatic SNVs and indels with Mutect2. BioRxiv [Preprint]. 2019. doi: https://doi.org/10.1101/861054
29. Van der Auwera GA, Carneiro M, Hartl C, et al. From FastQ data to high confidence variant calls: the Genome Analysis Toolkit best practices pipeline. Curr Protoc Bioinformatics. 2013;43(1110):11.10.1-11.10.33. doi: https://doi.org/10.1002/0471250953.bi1110s43
30. Kuilman T. CopywriteR: copy number information from targeted sequencing using off-target reads [Internet]. R package Version 2.26.0. [place unknown]: Bioconductor; c2003. doi: https://doi.org/10.18129/B9.bioc.CopywriteR
31. Danecek P, Bonfield JK, Liddle J, et al. Twelve years of SAMtools and BCFtools. Gigascience. 2021;10(2):giab008. doi: https://doi.org/10.1093/gigascience/giab008
32. McLaren W, Gil L, Hunt SE, et al. The ensembl variant effect predictor. Genome Biol. 2016;17(1):122. doi: https://doi.org/10.1186/s13059-016-0974-4
33. Conselho Nacional de Saúde (BR). Resolução nº 466, de 12 de dezembro de 2012. Aprova as diretrizes e normas regulamentadoras de pesquisas envolvendo seres humanos [Internet]. Diário Oficial da União, Brasília, DF. 2013 jun 13 [acesso 2022 mar 20]; Seção 1:59. Disponível em: https://bvsms.saude.gov.br/bvs/saudelegis/cns/2013/res0466_12_12_2012.html
34. Conselho Nacional de Saúde (BR). Resolução nº 510, de 7 de abril de 2016. Dispõe sobre as normas aplicáveis a pesquisas em Ciências Humanas e Sociais cujos procedimentos metodológicos envolvam a utilização de dados diretamente obtidos com os participantes ou de informações identificáveis ou que possam acarretar riscos maiores do que os existentes na vida cotidiana, na forma definida nesta Resolução [Internet]. Diário Oficial da União, Brasília, DF. 2016 maio 24 [acesso 2022 mar 20]; Seção 1:44. Disponível em: http://bvsms.saude.gov.br/bvs/saudelegis/cns/2016/res0510_07_04_2016.html
35. Landrum MJ, Lee JM, Benson M, et al. ClinVar: improving access to variant interpretations and supporting evidence. Nucleic Acids Res. 2018;46(D1):D1062-7. doi: https://doi.org/10.1093/nar/gkx1153
36. Clark K, Karsch-Mizrachi I, Lipman DJ, et al. GenBank. Nucleic Acids Res. 2016;44(D1):D67-72. doi: https://doi.org/10.1093/nar/gkv1276
37. Mehra S, Deshpande N, Nagathihalli N. Targeting PI3K pathway in pancreatic ductal adenocarcinoma: rationale and progress. Cancers (Basel). 2021;13(17):4434. doi: https://doi.org/10.3390/cancers13174434
38. Liu J, Kang R, Tang D. The KRAS-G12C inhibitor: activity and resistance. Cancer Gene Ther. 2021;29(7):875-878. doi: https://doi.org/10.1038/s41417-021-00383-9
39. Wijnen R, Pecoraro C, Carbone D, et al. Cyclin Dependent Kinase-1 (CDK-1) Inhibition as a Novel Therapeutic Strategy against Pancreatic Ductal Adenocarcinoma (PDAC). Cancers (Basel). 2021;13(17):4389. doi: https://doi.org/10.3390/cancers13174389
Recebido em 24/7/2022
Aprovado em 5/10/2022
Editor-associado: Claudio Gustavo Stefanoff. Orcid iD: https://orcid.org/0000-0001-7050-3269
Editora-científica: Anke Bergmann. Orcid iD: https://orcid.org/0000-0002-1972-8777
![]()
Este é um artigo publicado em acesso aberto (Open Access) sob a licença Creative Commons Attribution, que permite uso, distribuição e reprodução em qualquer meio, sem restrições, desde que o trabalho original seja corretamente citado.
©2019 Revista Brasileira de Cancerologia | Instituto Nacional de Câncer | Ministério da Saúde