ARTIGO ORIGINAL
Identificação de Genes Associados à Agressividade do Câncer Testicular
Identification of Genes Associated with Testicular Cancer Aggressiveness
Identificación de Genes Asociados con la Agresividad del Cáncer de Testículo
https://doi.org/10.32635/2176-9745.RBC.2024v70n2.4553
Graciele de Souza Medeiros1; Barbara Cardoso de Oliveira2; Vinícius Barbosa Parula Fernandes3; Vinicius Santos Cardoso4; Gabriel Arantes dos Santos5; Poliana Romão da Silva6; Sabrina Thalita dos Reis7
1-4Universidade Atenas Passos. Passos (MG), Brasil. E-mails: souzagra@hotmail.com; barabaraoliveiracardoso@gmail.com; viniciusbarbosaparula@gmail.com; viniciusantosmed@gmail.com. Orcid iD: https://orcid.org/0009-0004-5562-2007; Orcid iD: https://orcid.org/0000-0002-7502-2509; Orcid iD: https://orcid.org/0000-0002-9968-3619; Orcid iD: https://orcid.org/0000-0002-7132-9917
5-7Universidade de São Paulo Faculdade de Medicina. São Paulo (SP), Brasil. E-mails: arantes_gabriel@hotmail.com; romaosilva.poliana@gmail.com; sabrinareis@usp.br. Orcid iD: https://orcid.org/0000-0003-1602-9193; Orcid iD: https://orcid.org/0000-0003-0410-2349; Orcid iD: https://orcid.org/0000-0002-3564-3597
Endereço para correspondência: Graciele de Souza Medeiros. Rua Bartolomeu de Gusmão, 185, apto. 601 – Belo Horizonte. Passos (MG), Brasil. CEP 37900-026. E-mail: souzagra@hotmail.com
RESUMO
Introdução: Os tumores de células germinativas testiculares representam cerca de 97% dos cânceres testiculares. Histologicamente, classificam-se em seminomas e não seminomas, tendo aplicabilidade diagnóstica e prognóstica. O sucesso terapêutico depende do diagnóstico precoce associado ao correto estadiamento, sendo então de grande importância a avaliação de biomarcadores que possam contribuir para o manejo dessa doença. Objetivo: Identificar os genes que podem estar correlacionados com o prognóstico e a sobrevida no câncer testicular. Método: Análise de bioinformática utilizando 137 amostras de câncer testicular do The Cancer Genome Atlas e 165 amostras de tecido testicular normal do The Genotype-Tissue Expression. A identificação dos genes e análises subsequentes foram feitas pelo GEPIA2. Resultados: Inicialmente avaliou-se, em relação à expressão gênica, os 500 genes mais associados com a sobrevida global do câncer testicular e os 500 com a sobrevida livre de doença. Em seguida, foi realizada a sobreposição dessas duas listas e construído um diagrama de Venn mostrando os 13 genes em comum. Destes, mantiveram-se apenas os codificadores de proteína, verificando quais diferiram significativamente do tecido normal em relação à expressão gênica. Somente ATP10A, SAMD14 e PCAL4 mostraram diferença com significância estatística, todos subexpressos no câncer testicular. A análise deles em conjunto foi ainda mais significativa para a sobrevida global e livre de doença. Conclusão: Foram identificados nesta análise in silico três genes que demonstraram associação significativa de sua expressão com a sobrevida e o prognóstico dos pacientes com câncer testicular.
Palavras-chave: Neoplasias Testiculares; Células Germinativas; Biologia Computacional/estatística & dados numéricos; Prognóstico.
ABSTRACT
Introduction: Testicular germ cell tumors represent approximately 97% of testicular cancers. Histologically, they are classified into seminomas and non-seminomas, having diagnostic and prognostic applicability. Therapeutic success depends on early diagnosis associated with correct staging, the evaluation of biomarkers is important for the correct management of this disease. Objective: To identify genes that may be correlated with prognosis and survival in testicular cancer. Method: Bioinformatics analysis was performed using 137 testicular cancer samples from The Cancer Genome Atlas and 165 normal testicular tissue samples from The Genotype-Tissue Expression. Gene identification and subsequent analyzes were performed using GEPIA2. Results: Initially, in relation to gene expression, the 500 genes most significantly associated with overall survival from testicular cancer and the 500 with disease-free survival were evaluated. These two lists were then superimposed and a Venn diagram was constructed showing the 13 genes in common. Of these, only the protein-coding genes were kept, investigating which ones differed significantly from normal tissue in relation to gene expression. Only ATP10A, SAMD14 and PCAL4 showed a statistically significant difference, all of which were under-expressed in testicular cancer. The joint analysis of these genes was even more significant for overall and disease-free survival. Conclusion: Three genes were identified in the analysis in silico which demonstrated significative association of the expression with survival and prognosis of patients with testicular cancer.
Key words: Testicular Neoplasms; Germ Cells; Computational Biology/statistics & numerical data; Prognosis.
RESUMEN
Introducción: Los tumores de células germinales testiculares representan aproximadamente el 97% de los cánceres de testículo. Histológicamente se clasifican en seminomas y no seminomas, teniendo aplicabilidad diagnóstica y pronóstica. El éxito terapéutico depende de un diagnóstico temprano asociado a una correcta estadificación, siendo esta última altamente beneficiosa debido a los marcadores genéticos que indican cómo tratar la enfermedad. Objetivo: Identificar genes que puedan estar correlacionados con el pronóstico y la supervivencia en el cáncer testicular. Método: El análisis bioinformático se realizó utilizando 137 muestras de cáncer testicular de The Cancer Genome Atlas y 165 muestras de tejido testicular normal de The Genotype-Tissue Expression. La identificación de genes y los análisis posteriores se realizaron utilizando GEPIA2. Resultados: Inicialmente, en relación con la expresión génica, se evaluaron los 500 genes más significativamente asociados con la supervivencia global del cáncer testicular y los 500 con la supervivencia libre de enfermedad. Luego se superpusieron estas dos listas y se construyó un diagrama de Venn que muestra los 13 genes en común. De ellos, sólo se mantuvieron las codificantes de proteínas, comprobando cuáles diferían significativamente del tejido normal en relación con la expresión génica. Sólo ATP10A, SAMD14 y PCAL4 mostraron una diferencia estadísticamente significativa, todos los cuales estaban subexpresados en el cáncer testicular. El análisis conjunto de estos fue aún más significativo para la supervivencia general y libre de enfermedad. Conclusión: Los tres genes que se identificaron en este análisis in silico se expresan diferencialmente y demostraron una asociación significativa entre su expresión, la supervivencia y pronóstico de los pacientes con cáncer testicular.
Palabras clave: Neoplasias Testiculares; Células Germinativas; Biología Computacional/estadística & datos numéricos; Pronóstico.
INTRODUÇÃO
O câncer testicular (CT) é o tumor mais comum em homens com idade entre 15 e 34 anos1. Ele pode se apresentar como tumores de células de leydig, células de sertoli, células da granulosa que geralmente são benignos, mas têm potencial maligno, além dos tumores de células germinativas testiculares (TCGT) que são malignos e correspondem a 95% dos casos de CT2. Histologicamente, o TCGT pode ser classificado em seminomas e não seminomas. Essa distinção histológica é altamente relevante para a etiologia e tratamento do tumor, pois os tumores não seminomas são mais propensos à metástase1.
Os fatores de risco que envolvem o CT estão relacionados à criptorquidia – condição que ocasiona a não descida do testículo –, história familiar positiva de CT, etnia, idade e infertilidade1.
São consideradas neoplasias raras, com incidência estimada em cerca de 5 a cada 100 mil indivíduos3. Geralmente, o CT possui caráter agressivo, em especial para tumores não seminomatosos, porém possui alta taxa de cura com as terapias atuais4-6, com taxa de mortalidade próxima a 0,26/100 mil no Brasil, mostrando tendência para o crescimento nos últimos anos7.
Atualmente, para o diagnóstico de CT utilizam-se marcadores tumorais séricos e exames de imagens, ao apresentar massa testicular recomenda-se que seja feita a ultrassonografia testicular e, em seguida, uma pesquisa de marcadores tumorais séricos, sendo eles α-fetoproteína (AFP), gonadotrofina coriônica humana (hCG) e lactato desidrogenase (LDH). Porém, esses marcadores ainda apresentam baixa sensibilidade específica para o TCGT, sendo necessários novos estudos com a finalidade de encontrar biomarcadores que possam auxiliar no manuseio do CT7.
Assim como em outras neoplasias, o sucesso terapêutico depende do diagnóstico precoce associado ao correto estadiamento4-6,8, que pode ser aprimorado pela adição de marcadores moleculares. A partir disso, estudos buscam marcadores específicos para seminomas e biomarcadores que possam auxiliar na estratificação de risco relacionada ao estadiamento clínico, e na orientação de escolha e monitorização terapêuticas, principalmente na doença avançada9. Com base no exposto, o objetivo desse estudo foi identificar os genes que podem estar associados com o prognóstico e a sobrevida no CT.
MÉTODO
Neste trabalho, foi realizada a análise de bioinformática utilizando 137 amostras de CT do The Cancer Genome Atlas (TCGA)10 e 165 amostras de tecido testicular normal do The Genotype-Tissue Expression (GTEx)11. Por se tratar de uma análise in silico, não foi necessário o parecer do Comitê de Ética em Pesquisa (CEP) para a realização desta pesquisa, conforme a Resolução n.º 51012/2016 do Conselho Nacional de Saúde (CNS).
Para a seleção de genes que estão associados à sobrevida, foram utilizados dados disponibilizados na ferramenta GEPIA213 com genes da seleção de conjunto de dados do TCGA, como método de sobrevida global, e, em seguida, foi feita a sobrevida livre de doença empregando a mediana como corte. Na matriz de expressão dentro dessa ferramenta, a densidade de cor em cada bloco representa o valor de expressão mediana de um gene em um determinado tecido, normalizado pelo valor de expressão mediana máximo em todos os blocos. Nessa etapa, foram selecionados os primeiros 500 genes com diferença estatística significativa, ou seja, aqueles que tinham o menor valor de p. As curvas de sobrevida global e sobrevida livre de doença foram realizadas nessa mesma ferramenta utilizando a mediana de expressão dos genes como corte.
As análises da associação da expressão gênica, presença de metilação com o tipo histológico, idade, raça e estádio da doença foram realizadas pela ferramenta Ualcan14 utilizando como consulta “TCGA” seguido de “TCGT - tumor de células germinativas testiculares” e, logo após, a expressão correlacionada a “Tipo histológico”, “Idade”, “Raça”, “Estágios individuais do câncer” e “Metilação” de cada gene.
A avaliação das alterações genéticas em cada gene foi realizada pela ferramenta cBioportal15 utilizando “TESTICULAR” e, em seguida, “TCGA, Firehose Legacy” como consulta. Realizando, assim, a análise de mutações e supostas alterações no número de cópias de GISTIC de cada gene.
Inicialmente, foram selecionados 100 genes com padrão de expressão similar ao ATP10A, HPCAL4 e SAMD14 no CT pela ferramenta GEPIA213. Então, foi utilizada a ferramenta WebGestalt16 para a análise de enriquecimento, tendo como parâmetros básicos o organismo de interesse: Homo sapiens, banco de dados funcional: geneontologia e método de interesse: “ORA sample run” e pathway + KEGG de cada lista.
RESULTADOS
Para selecionar os genes diferencialmente expressos no CT, utilizou-se a plataforma GEPIA2. Foram selecionados os 500 primeiros genes associados com a sobrevida global do CT e os 500 genes associados com a sobrevida livre de doença. Posteriormente, foi realizada a sobreposição dessas duas listas e construído um diagrama de Venn com o objetivo de selecionar os genes que estavam associados tanto com a sobrevida global quanto com a sobrevida livre da doença. Nesta análise, foram encontrados 13 genes associados significativamente com as duas sobrevidas avaliadas: SAMD14, CISD3, HPCAL4, RP11-442G21.2, RN7SL208P, ATP10A, CASC8, HOXA9, ANPEP, CTC-459F4.9, RPL35P1, TMLHE-AS1 e ARPC3P1. Após a análise de cada um desses genes, selecionaram-se aqueles que eram codificadores de proteínas resultando em ATP10A, SAMD14, HPCAL4, CISD3, HOXA9, ANPEP. Para escolher quais genes seriam os mais relevantes no CT, foi avaliada a expressão desses seis genes ainda empregando a plataforma GEPIA2 nas amostras de tumores testiculares e comparando com as amostras de testículo normal disponíveis na plataforma. Três genes se mostraram diferencialmente expressos com significância estatística (p < 0,05) nos tumores testiculares – ATP10A, SAMD14 e HPCAL4 (Figura 1). Todos os genes estavam subexpressos no CT quando comparados ao controle obtido no GTEx.
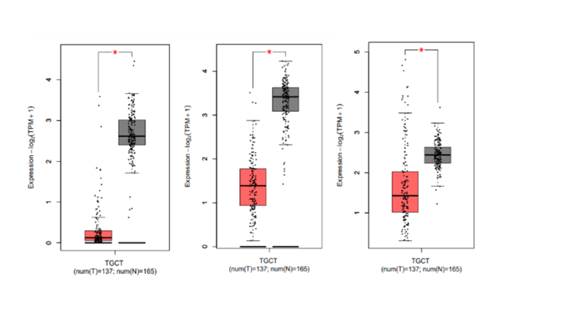 |
Figura 1. Análise comparativa dos níveis de expressão log2(TPM+1) dos genes diferencialmente expressos com significância estatística (p < 0,05), HPCAL4, ATP10A e SAMD14 respectivamente, entre 137 amostras de tecido de câncer testicular (vermelho) e 165 amostras controle (cinza)
Legendas: TCGT = tumores de células germinativas testiculares; TPM = amostras transcritas por milhão.
Ainda foram analisadas se as expressões desses três genes estavam associadas aos dados de sobrevida. Essa análise em conjunto foi capaz de predizer de modo significativo a sobrevida global (p = 0,046) e a sobrevida livre de doença (p = 0,013) (Figura 2).
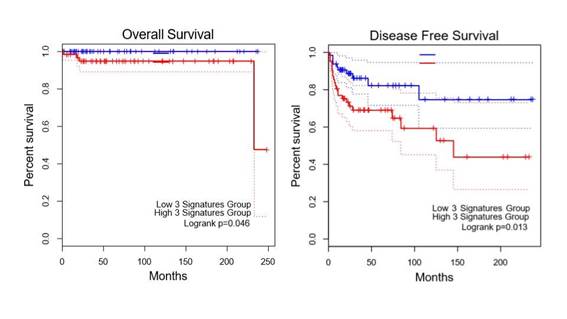 |
Figura 2. Análise do percentual de sobrevivência (percent survival), de 0,0 a 1,0, de acordo com a expressão conjunta dos três genes (HPCAL4, ATP10A e SAMD14) com baixa assinatura (azul) e alta assinatura (vermelho) ao longo dos meses (Months), de 0 a 250 meses, na sobrevida global (overall survival) e sobrevida livre de doença (disease free survival)
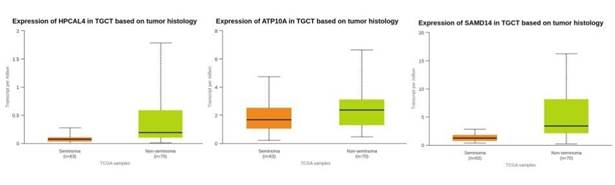 |
Figura 3. Análise da expressão gênica em amostras de tumores de células germinativas testiculares, de HPCAL4, APT10A e SAMD14 respectivamente, com base na histologia tumoral em seminomas (laranja) com 63 amostras e não seminomas (verde) com 70 TPM. HPCAL4 teve variação de 0 a 2 em TPM e p = 2,81900E-03; ATP10A variação de 0 a 8 em TPM e p = 4,523700E-03; SAMD14 variação de 0 a 20 em TPM e p = 8,26320001312553E-09
Legendas: TCGT = tumores de células germinativas testiculares; TPM = amostras transcritas por milhão.
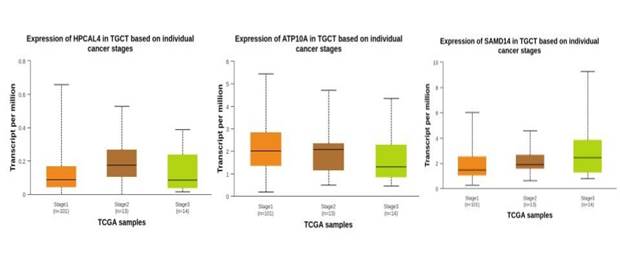 |
Figura 4. Análise da expressão gênica em amostras de tumores de células germinativas testiculares de HPCAL4, ATP10A e SAMD14, respectivamente, com base na histologia tumoral do estádio 1 (laranja) com n =101, estádio 2 (marrom) com n = 13 e estádio 3 (verde) com n = 14, transcrito por milhão. No gráfico do gene HPCAL4, houve variação de 0 a 0,8 do TPM, com estádio 1 vs. estádio 2 de p = 1,243810E-01, estádio 1 vs. estádio 3 de p = 6,883500E-02, estádio 2 vs. estádio 3, p = 6,771800E-01. No ATP10A, a variação foi de 0 a 6 TPM, com estádio 1 vs. estádio 2 de p = 6,509200E-01, estádio 1 vs. estádio 3 de p = 8,612300E-02, estádio 2 vs. estádio 3 p = 4,228200E-01. No SAMD14, a variação foi de 0 a 10 TPM, com estádio 1 vs. estádio 2 de p = 9,889800E-01, estádio 1 vs. estádio 3 de p = 3,994600E-01, estádio 2 vs. estádio 3, p = 5,148800E-01
Legendas: TCGT = tumores de células germinativas testiculares; TCGA samples = amostras The Cancer Genome Atlas; TPM = amostras transcritas por milhão.
Realizou-se uma comparação da expressão dos três genes entre tumores testiculares do tipo histológico seminoma e não seminoma e foi observado que a expressão dos três genes foi maior no tipo histológico não seminoma (Figura 3).
Ao avaliar a expressão dos genes com o estádio tumoral, observou-se expressões maiores de cada gene associadas a um estádio específico da doença, o gene ATP10A foi mais expresso em tumores T1, HPCAL4, em tumores T2 e SAMD14, em tumores T3 (Figura 4).
Ainda foi avaliada a expressão desses genes em diferentes etnias e faixas etárias, demonstrou-se que em relação à raça, o gene ATP10A teve maior expressão em asiáticos (comparação de caucasiano vs. afro-americanos com p = 2,857400E-01, caucasianos vs. asiáticos, p = 8,720400E-01, e afro-americanos vs. asiáticos p = 2,466800E-01); o SAMD14 teve maior expressão em caucasianos e afro-americanos (comparação de caucasiano vs. afro-americanos com p = 5,935400E-01, caucasianos vs. asiáticos, p = 7,486800E-01, e afro-americanos vs. asiáticos, p = 9,479600E-01); e o HPCAL4 não mostrou diferença entre elas (comparação de caucasiano vs. afro-americanos com p = 3,880300E-03, caucasianos vs. asiáticos, p = 9,851800E-01, e afro-americanos vs. asiáticos p = 3,747200E-01).
Na análise entre faixas etárias, o ATP10A teve expressão inversamente proporcional a idade, ou seja, apresentou maior expressão na faixa etária de 21 a 40 anos e menor expressão em 61 a 80 anos (p = 3,420400E-01), o SAMD14 mostrou-se com maior expressão entre 61 e 80 anos (p = 4,895400E-01) e no HPCAL4 não foi observado diferença significante (comparação de 21-40 a vs. 41-60a com p = 7,732200E-02, 21-40a vs. 61-80ª, p = 1,437890E-03, e 41-60a vs. 61-80a, p = 7,543600E-01).
Posteriormente, foi realizada a análise de mutações por presença de metilação. Para os genes ATP10A (p < 1E-12) e HPCAL4 (p = 6,66344757149773E-12), não foi demonstrada mutação, já ao avaliar o gene SAMD14 (p = 5,028100E-02), apenas um paciente, ou seja, 0,7% da amostra apresentava uma mutação de alteração de nucleotídeo único do tipo missense (G397W).
E, por último, foi realizada análise de enriquecimento do conjunto de 100 genes com expressão similar à de ATP10A, HPCAL4 e SAMD14 para avaliação e compreensão da função expressa de cada agrupamento e consequentemente desses três genes (Quadro 1).
Quadro 1. Representação das funções associadas a conjuntos de genes com expressão similar à de ATP10A, HPCAL4 e SAMD14 no câncer testicular, identificados por meio da análise de enriquecimento utilizando a metodologia 'Ora sample run' e 'Pathway + KEGG'
|
Método |
ATP10A |
HPCAL4 |
SAMD14 |
|
Ora sample run |
Motilidade celular dependente de cílio ou flagelo, motilidade espermática, fusão célula-célula, formação de sincício, fertilização, formação de feixe de microtúbulos, processo celular envolvido na reprodução em organismo multicelular, movimento baseado em microtúbulos, armazenamento de cílio |
Motilidade celular dependente de cílio ou flagelo, formação de feixe de microtúbulos, migração de neurônios, via de sinalização do receptor de glutamato, transmissão sináptica, glutamatérgico, desenvolvimento do núcleo neural, polimerização ou despolimerização de microtúbulos, regulação do receptor de neurotransmissor de membrana pós-sináptica l, motilidade espermática, regulação do receptor de neurotransmissor atividade, organização de sinapses, diferenciação de células fotorreceptoras, localização de proteínas na sinapse, regulação negativa do desenvolvimento do sistema nervoso, desenvolvimento do rombencéfalo, regulação da sinalização transsináptica, comportamento adulto, desenvolvimento do pâncreas, regulação da estrutura ou atividade da sinapse, transmissão sináptica química, pós-sináptica, regulação do processo baseado em microtúbulos, diferenciação de neurônios do sistema nervoso central, ciclo de vesículas sinápticas, exocitose regulada por íons de cálcio, organização de projeção de neurônios, movimento baseado em microtúbulos, transporte mediado por vesículas na sinapse, desenvolvimento de dendritos, comportamento locomotor, regulação negativa do desenvolvimento de chamadas, regulação da resposta ao medicamento, diferenciação de células epiteliais colunares/cuboidais, desenvolvimento do prosencéfalo, cognição, regulação do processo do sistema neurológico, orientação da projeção de neurônios, organização do cílio, regulação da atividade do transportador, transporte de neurotransmissores, desenvolvimento de axônios, regulação negativa da organização da projeção celular, regulação do desenvolvimento da projeção de neurônios, gliogênese, localização dentro da membrana, regulação do potencial de membrana, sinalização do organismo multicelular, maturação do desenvolvimento, regulação positiva do transporte de íons, crescimento do desenvolvimento envolvido na morfogênese, localização de vesículas, comprometimento do destino celular, transporte de íons potássio, regulação dos níveis de neurotransmissores , liberação de sinal, desenvolvimento do sistema sensorial, regulação positiva da neurogênese |
Adesão de células II via adesão molecular à membrana plasmática
|
|
Pathway + KEGG |
- |
Sinapse glutamatérgica
|
Migração transendotelial de leucócitos, hepatite C, moléculas de adesão celular, junção estreita
|
Nota: No quadro, estão listadas apenas as funções com significância estatística (p ≤ 0,05).
DISCUSSÃO
O CT é um desafio clínico17, visto que representa uma das neoplasias mais comuns que acometem o sexo masculino, que inclui os TCGT17. O CT é classificado em dois grupos principais: seminomas e não seminomas18, e, somado a isso, sabe-se que o surgimento do CT é dependente de fatores genéticos13. Dessa forma, muitos estudos estão sendo realizados para desenvolver novos biomarcadores moleculares tumorais com maior especificidade e sensibilidade19. Contudo, no cenário atual, há uma quantidade considerada pequena de biomarcadores conhecidos considerando o CT. Diante disso, neste estudo, realizou-se uma análise de bioinformática em busca de biomarcadores em que foram encontrados três genes, os quais se mostraram diferencialmente expressos com significância estatística e associados à agressividade da neoplasia testicular. Os genes diferencialmente expressos foram: ATP10A, SAMD14 e HPCAL4, todos subexpressos no CT.
É relevante salientar que há uma mínima disponibilidade de estudos os quais sustentam a base genética do CT17, portanto, há poucos biomarcadores documentados para o prognóstico. Os genes codificadores de proteínas ATP10A, SAMD14 e HPCAL4 encontrados neste trabalho estão diferencialmente expressos no CT e, ainda, demonstram uma relação associativa de sua expressão com a taxa de sobrevida. Nesse cenário, a relevância deste estudo justifica-se por esses genes ainda não terem sido avaliados na literatura como marcadores moleculares do CT19.
Sabe-se que os marcadores tumorais AFP, bHCG e LDH são importantes no manejo clínico de TCGT20. Contudo, a expressão é relativamente baixa em TCGT, apenas 60% dos CT demonstram elevação dessas moléculas no diagnóstico inicial, o que torna sua aplicabilidade clínica com baixa especificidade e deixam a desejar no acompanhamento e monitoramento da doença avançada. Diante disso, são necessários biomarcadores mais específicos para o TCGT, uma vez que eles são essenciais para orientar a escolha do tratamento, principalmente quando a doença já está em estádio avançado9.
Ao analisar os três genes identificados neste trabalho, observou-se que predominantemente há uma maior expressão gênica nos tipos não seminomas. Sabe-se que existe uma expressão diferencial a depender do subtipo histológico e que os tumores de células germinativas não seminomatosas (NSGCT) são os mais prováveis de causar metástases21, o que acaba por impactar na qualidade da sobrevida da doença.
Sabe-se também que o CT, normalmente, é mais comum em homens na faixa etária de 15 a 34 anos9, o que condiz com os achados deste estudo, já que foi identificado maior expressão dos genes ATP10A e HPCAL4 entre as idades de 21 a 40 anos, responsáveis respectivamente por maior expressão nos estádios 1 e 2. Porém, houve uma discrepância em relação ao gene SAMD14 que foi encontrado com maior expressão na faixa etária de 61 a 80 anos, podendo estar associado ao estádio avançado da doença, visto que apresentou maior expressão no estádio 3. Esses achados podem nortear novas pesquisas para confirmação da correlação da expressão desses genes com um estádio específico da doença em busca de comprovação de biomarcadores com alto potencial de classificação prognóstica. Análises mostraram também a expressão desses genes em relação à raça: o ATP10A é mais expresso em asiáticos, o HPCAL4 em afro-americanos e asiáticos e o SAMD14 em caucasianos e afro-americanos. Vale ressaltar que na literatura foi documentado que a incidência de TCGT varia entre as populações, sendo maior em indivíduos de ascendência europeia em comparação com os africanos22.
Quanto aos genes, o ATP10A foi identificado como responsável por produzir uma proteína do tipo flipase, a qual transporta fosfatidilcolina do folheto externo para o folheto interno da membrana plasmática8. Assim, identifica-se no presente estudo que o gene é diferencialmente expresso em tumores não seminomatosos, visto uma hipometilação do seu sítio promotor, não havendo diferenças significativas ao se comparar idade, raça ou estádios. Diante disso, ao realizar uma análise de enriquecimento para avaliar as vias de atuação gênicas, observou-se que existe uma grande atuação na via de fusão célula-célula e formação do sincício. Sabe-se que a desregulação da adesão celular pode desempenhar um papel no processo de transformação, contribuindo para processos metastáticos13. Logo, é possível inferir que a hiperexpressão do gene ATP10A pode implicar uma menor adesão entre as células e, assim, uma maior capacidade de metástase das células neoplásicas, validando os achados citados.
Sabe-se que o SAMD14 foi identificado em mastócitos de espécimes de câncer de próstata como mediador da comunicação intercelular e interação epitelial no microambiente e que um microambiente favorável, feito a partir da comunicação bidirecional entre células e a matriz extracelular (MEC), é essencial para o desenvolvimento da tumorigênese e metástase13. Os resultados do presente estudo podem ser correlacionados com tal afirmação, uma vez que, ao realizar a análise de enriquecimento, o SAMD14 apresentou como função a adesão de células-II via adesão molecular à membrana plasmática. Além disso, pacientes com câncer gástrico que apresentam baixa expressão e hipermetilação do promotor de SAMD14 têm pior sobrevida global23, o que pode ser relacionado com os achados deste estudo, no qual foi observada a subexpressão do gene no TCGT quando comparado ao tecido normal. Estudos recentes têm mostrado uma terapêutica para linfoma primário do sistema nervoso central em forma de anticorpo pela integração do antígeno receptor de células B neurabin-I/SAMD1424, podendo ser uma direção promissora de tratamento para TCGT por meio de novas pesquisas, fundamentadas nos resultados apresentados nesses estudos e achados do presente trabalho.
CONCLUSÃO
Os três genes codificadores de proteína estão diferencialmente expressos no CT, no tipo histológico não seminoma, e demonstraram associação significativa de sua expressão com a sobrevida e consequentemente com o prognóstico dos pacientes, sendo potenciais biomarcadores para o CT. No entanto, mais estudos detalhados ainda são necessários para a validação desses genes no CT.
CONTRIBUIÇÕES
Todos os autores contribuíram substancialmente na concepção e no planejamento do estudo; na obtenção, análise e interpretação dos dados; na redação e revisão crítica; e aprovaram a versão final a ser publicada.
DECLARAÇÃO DE CONFLITO DE INTERESSES
Nada a declarar.
FONTES DE FINANCIAMENTO
Não há.
REFERÊNCIAS
1. Baird DC, Meyers GJ, Hu JS. Testicular cancer: diagnosis and treatment. Am Fam Physician. 2018;97(4):261-8.
2.Smith ZL, Werntz RP, Eggener SE. Testicular cancer. Med Clin North Am. 2018;102(2):251-64. doi: https://doi.org/10.1016/j.mcna.2017.10.003
3.Teng LK, Pereira BA, Keerthikumar S, et al. Mast Cell-Derived SAMD14 is a novel regulator of the human prostate tumor microenvironment. Cancers. 2021;13(6):1237. doi: https://doi.org/10.3390/cancers13061237
4.Miller KD, Nogueira L, Mariotto AB, et al. Cancer treatment and survivorship statistics, 2019. CA. 2019;69(5):363-85. doi: https://doi.org/10.3322/caac.21565
5.Mourão TC, Curado MP, Oliveira RAR, et al. Epidemiology of urological cancers in Brazil: trends in mortality rates over more than two decades. J Epidemiol Glob Health 2022;12(3):239-47. doi: https://doi.org/10.1007/s44197-022-00042-8
6. Kliesch S, Schmidt S, Wilborn D et al. Management of germ cell tumours of the testis in adult patients. german clinical practice guideline part i: epidemiology, classification, diagnosis, prognosis, fertility preservation, and treatment recommendations for localized stages. Urol Int. 2021;105(3-4):1-12. doi: https://doi.org/10.1159/000510407
7. Yang J, Yang Q. Identification of core genes and screening of potential targets in glioblastoma multiforme by integrated bioinformatic analysis. Front Oncol. 2021;10(615976):1-12. doi: https://doi.org/10.3389/fonc.2020.615976
8. Souza KW, Reis PE, Gomes IP, et al. Estratégias de prevenção para câncer de testículo e pênis: revisão integrativa. Rev Esc Enferm USP. 2011;45(1):277-82. doi: https://doi.org/10.1590/s0080-62342011000100039
9. Leão R, Ahmad AE, Hamilton RJ. Testicular cancer biomarkers: a role for precision medicine in testicular cancer. Clin Genitourin Cancer. 2019;17(1):e176-83. doi: https://doi.org/10.1016/j.clgc.2018.10.007
10. Nacional Cancer Institute, Center for Cancer Genomics [Internet]. Annapolis: NIH; 2006. The Cancer Genome Atlas (TCGA); 2006. [acesso 2024 mar 8]. Disponível em: https://www.cancer.gov/ccg/research/genome-sequencing/tcga
11. GTEx: The Genotype-Tissue Expression [Internet]. Versão phs000424.v9.p2. Bethesda: NIH; [sem data]. [acesso 2024 mar 8]. Disponível em: https://gtexportal.org/home/
12. Conselho Nacional de Saúde (BR). Resolução n° 510, de 7 de abril de 2016. Dispõe sobre as normas aplicáveis a pesquisas em Ciências Humanas e Sociais cujos procedimentos metodológicos envolvam a utilização de dados diretamente obtidos com os participantes ou de informações identificáveis ou que possam acarretar riscos maiores do que os existentes na vida cotidiana, na forma definida nesta Resolução [Internet]. Diário Oficial da União, Brasília, DF. 2016 maio 24 [acesso 2024 jan 20]; Seção I:44. Disponível em: http://bvsms.saude.gov.br/bvs/saudelegis/cns/2016/res0510_07_04_2016.html
13. Tang Z, Kang B, Li C, et al. GEPIA2: an enhanced web server for large-scale expression profiling and interactive analysis. Nucleic Acids Res. 2019;47(W1):W556-60. doi: https://doi.org/10.1093/nar/gkz430
14. Chandrashekar DS, Karthikeyan SK, Korla PK, et al. UALCAN: uma atualização para a plataforma integrada de análise de dados de câncer. Neoplasia. 2022;25:18-27. doi: https://doi.org/10.1016/j.neo.2022.01.001
15. Bruijn I, Kundra R, Mastrogiacomo B, et al. Analysis and visualization of longitudinal genomic and clinical data from the AACR project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023;83(23):3861-7. doi: https://doi.org/10.1158/0008-5472.can-23-0816
16. WebGestalt [Internet]. Versão 1.2. Houston: Zhang Lab; ©2024. [acesso 2024 mar 8]. Disponível em: http://webgestalt.org/
17. Toni L, Šabovic I, Cosci I, et al. Testicular cancer: genes, environment, hormones. Front Endocrinol. 2019;10(408):1-11. doi: https://doi.org/10.3389/fendo.2019.00408
18. Boccellino M, Vanacore D, Zappavigna S, et al. Testicular cancer from diagnosis to epigenetic factors. Oncotarget. 2017;8(61):104654-63. doi: https://doi.org/10.18632/oncotarget.20992
19. Chieffi P, Franco R, Portella G. Molecular and cell biology of testicular germ cell tumors. Int Rev Cell Mol Biol. 2009;278:277-308. doi: https://doi.org/10.1016/s1937-6448(09)78006-2
20. Murray MJ, Huddart RA, Coleman N. The present and future of serum diagnostic tests for testicular germ cell tumours. Nat Rev Urol. 2016;13(12):715-25. doi: https://doi.org/10.1038/nrurol.2016.170
21.Nauman M, Leslie SW. Nonseminomatous testicular tumors. In: StatPearls [Internet]. Treasure Island: StatPearls Publishing; 2023. [acesso 2024 mar 8]. Disponível em: https://www.ncbi.nlm.nih.gov/books/NBK568754/
22. Baylin SB, Jones PA. A decade of exploring the cancer epigenome — biological and translational implications. Nat Rev Cancer. 2011;11(10):726-34. doi: https://doi.org/10.1038/nrc3130
23.Chung CC, Kanetsky PA, Wang Z, et al. Meta-analysis identifies four new loci associated with testicular germ cell tumor. Nat Genet. 2013;45(6):680-5. doi: https://doi.org/10.1038/ng.2634
24. Xu X, Chang X, Xu Y et al. SAMD14 promoter methylation is strongly associated with gene expression and poor prognosis in gastric cancer. Int J Clin Oncol [Internet]. 21 mar 2020;25(6):1105-14. doi: 10.1007/s10147-020-01647-4
Recebido em 31/1/2024
Aprovado em 4/6/2024
Editor-associado: Claudio Gustavo Stefanoff. Orcid iD: https://orcid.org/0000-0001-7050-3269
Editora-científica: Anke Bergmann. Orcid iD: https://orcid.org/0000-0002-1972-8777
![]()
Este é um artigo publicado em acesso aberto (Open Access) sob a licença Creative Commons Attribution, que permite uso, distribuição e reprodução em qualquer meio, sem restrições, desde que o trabalho original seja corretamente citado.