REVISÃO DE LITERATURA
Utilização de DNA Tumoral Circulante na Análise de Prognóstico de Pacientes com Tumores Malignos Sólidos do Trato Gastrintestinal: Revisão Sistemática
Use of Circulating Tumor DNA in Prognostic Analysis of Patients with Solid Malignant Tumors of the Gastrointestinal Tract: Systematic Review
Uso del ADN Tumoral Circulante en el Análisis pronóstico de Pacientes con Tumores Malignos Sólidos del Tracto Gastrointestinal: Revisión Sistemática
https://doi.org/10.32635/2176-9745.RBC.2024v70n4.4873
Gabriel dos Santos Martins de Abreu1; Mariana
Albuquerque de Brito2; Ana Paula de Souza Ramos3
1-3Universidade Estadual do Sudoeste da Bahia (Uesb). Jequié (BA), Brasil.
1E-mail: gabrielsmabreu@gmail.com. Orcid iD: https://orcid.org/0009-0000-4270-2519
2E-mail: marianaalbuquerquebrito50@gmail.com. Orcid iD: https://orcid.org/0000-0002-4101-464X
3E-mail: aninharamos.bio@gmail.com. Orcid iD: https://orcid.org/0000-0002-5805-9596
Endereço para correspondência: Gabriel dos Santos Martins de Abreu. Av. José Moreira Sobrinho, S/N – Jequiezinho. Jequié (BA), Brasil. CEP 45205-490. E-mail: gabrielsmabreu@gmail.com
RESUMO
Introdução: O DNA tumoral circulante (ctDNA), um dos principais expoentes da biópsia líquida, constitui uma ferramenta promissora na área da oncologia. Contudo, seu uso na prática clínica, apesar de variado, necessita de maiores embasamentos. Objetivo: Avaliar o impacto da utilização do ctDNA como ferramenta para qualificar o prognóstico de pacientes com tumores malignos sólidos do trato gastrintestinal. Método: Revisão sistemática baseada em estudos do tipo coorte, utilizando as bases de dados MEDLINE, LILACS, SciELO, Science Direct e BASE. A avaliação da qualidade metodológica foi feita pela Newcaslte-Ottawa Quality Assessment Scale. Resultados: Dos 557 artigos encontrados inicialmente, a amostra final do presente estudo contou com 13 artigos. Observaram-se maiores taxas de recidiva tumoral e menores taxas de sobrevida em pacientes ctDNA-positivos com tumores de todos os principais sítios do trato gastrintestinal, em relação àqueles ctDNA-negativos. Essa correlação foi consistente ao longo de todos os estádios tumorais. Ademais, o ctDNA se mostrou mais eficaz na predição de recidiva tumoral comprovada por exame radiológico quando comparado ao antígeno carcinoembrionário, tipicamente utilizado nesse contexto. Conclusão: Os resultados encontrados apoiam a utilização do ctDNA no cenário descrito, de modo complementar às ferramentas de avaliação de prognóstico comumente utilizadas na prática clínica atual.
Palavras-chave: Biópsia líquida/métodos; DNA Tumoral Circulante; Prognóstico; Trato gastrintestinal/patologia.
ABSTRACT
Introduction: The circulating tumor DNA (ctDNA), one of the main exponents of liquid biopsy, constitutes a promising tool in the field of oncology. However, its use in clinical practice, despite being varied, requires further support. Objective: To evaluate the impact of using ctDNA as a tool to qualify the prognosis of patients with solid malignant tumors of the gastrointestinal tract. Method: Systematic review based on cohort studies, using the MEDLINE, LILACS, SciELO, Science Direct and BASE databases. The assessment of methodological quality was carried out by the Newcastle-Ottawa Quality Assessment Scale. Results: Of the 557 articles initially found, the final sample of the present study included 13 articles. Higher rates of tumor recurrence and lower survival rates were observed in ctDNA-positive patients with tumors from all main sites of the gastrointestinal tract, compared to those who were ctDNA-negative. This correlation was consistent across all tumor stages. Furthermore, ctDNA proved to be more effective in predicting tumor recurrence proven by radiological examination when compared to carcinoembryonic antigen, typically used in this context. Conclusion: The results found support the use of ctDNA in the described scenario, in a complementary way to the prognostic assessment tools commonly used in current clinical practice.
Key words: Liquid biopsy/methods; Circulating Tumor DNA; Prognosis; Gastrointestinal Tract/pathology.
RESUMEN
Introducción: El ADN tumoral circulante (ADNct), uno de los principales exponentes de la biopsia líquida, constituye una herramienta prometedora en el campo de la oncología. Sin embargo, su uso en la práctica clínica, a pesar de ser variado, requiere mayor sustento. Objetivo: Evaluar el impacto del uso del ADNct como herramienta para calificar el pronóstico de pacientes con tumores sólidos malignos del tracto gastrointestinal. Método: Revisión sistemática basada en estudios de cohortes, utilizando las bases de datos MEDLINE, LILACS, SciELO, Science Direct y BASE. La evaluación de la calidad metodológica se realizó mediante la Newcastle-Ottawa Quality Assessment Scale. Resultados: De los 557 artículos encontrados inicialmente, la muestra final del presente estudio incluyó 13 artículos. Se observaron tasas más altas de recurrencia tumoral y tasas de supervivencia más bajas en pacientes con ADNct positivo con tumores de todos los sitios principales del tracto gastrointestinal, en comparación con aquellos con ADNct negativo. Esta correlación fue consistente en todas las etapas del tumor. Además, el ADNct demostró ser más eficaz para predecir la recurrencia del tumor verificada mediante examen radiológico en comparación con el antígeno carcinoembrionario, típicamente utilizado en este contexto. Conclusión: Los resultados encontrados apoyan el uso de ADNct en el escenario descrito, de forma complementaria a las herramientas de evaluación pronóstica habitualmente utilizadas en la práctica clínica actual.
Palabras clave: Biopsia líquida/métodos; ADN Tumoral Circulante; Pronóstico; Tracto Gastrintestinal/patología.
No ano de 2022, cerca de 20 milhões de casos de câncer e 9,7 milhões de óbitos pela doença foram registrados no nível global1. No Brasil, vivencia-se um cenário semelhante, com mais de 16% das mortes ocorrendo em função de tumores malignos2.
Esse cenário epidemiológico pode ser parcialmente compreendido pela ampla exposição da população a fatores de risco determinantes para o surgimento de doenças, tais como agentes químicos, físicos e biológicos com potencial carcinogênico. Além disso, a gradual inversão da pirâmide etária, resultante das melhorias contínuas nos setores de saúde, farmacologia e infraestrutura, tem proporcionado uma crescente expectativa de vida. Embora esse fenômeno seja essencialmente positivo, sabe-se que a idade avançada constitui um dos principais fatores de risco para diversos tipos de câncer2.
Diante disso, ao passo em que o campo da oncologia cresce de forma expressiva nas últimas décadas, busca-se cada vez mais compreender os processos fisiopatológicos que levam à malignidade, bem como diferentes formas de rastreio, diagnóstico, avaliação de prognóstico e tratamento da doença. Nesse cenário, os biomarcadores configuram-se como uma ferramenta poderosa de avaliação do paciente oncológico, sendo capaz de driblar algumas das maiores dificuldades dos principais métodos ainda utilizados, como as biópsias tradicionais3.
No espectro dos biomarcadores, a biópsia líquida tem ganhado destaque em razão de seu potencial promissor na avaliação de pacientes oncológicos em todas as etapas supracitadas. Essa técnica não invasiva permite não apenas a análise das células tumorais circulantes (CTC), mas também a avaliação do DNA tumoral circulante (ctDNA). O ctDNA representa uma das principais vertentes desse procedimento, capaz de correlacionar a atividade da doença com os níveis de moléculas de DNA derivadas de células tumorais presentes na circulação sanguínea, em virtude de uma variedade de mecanismos bioquímicos4.
A utilização do ctDNA como ferramenta de avaliação oncológica revela-se particularmente útil nos casos dos tumores do trato gastrintestinal (TGI). Para além da alta relevância epidemiológica, manifestada por elevadas taxas de incidência e mortalidade no Brasil e no mundo, os tumores do TGI são de difícil detecção precoce, em virtude de sua sintomatologia escassa e inespecífica em seus estágios iniciais5.
É importante destacar que a acurácia dessa correlação não é uniforme entre diferentes tipos de tumores e nas várias fases da avaliação oncológica. Portanto, torna-se evidente a necessidade de realizar estudos adicionais com o objetivo de esclarecer as aplicações mais eficazes dessa tecnologia. Objetiva-se com este estudo avaliar o impacto da utilização de ctDNA como ferramenta para qualificar o prognóstico de pacientes com tumores malignos sólidos do trato gastrintestinal.
MÉTODO
Revisão sistemática da literatura, cuja base para a construção da escrita constituiu-se do protocolo Preferred Reporting Items for Systematic Reviews and Metanalyses (PRISMA)6. Essa revisão sistemática foi registrada na base International Prospective Register of Systematic Reviews (PROSPERO)7, identificada pelo número CRD42023460611.
Por se tratar de um estudo de prognóstico, a estratégia adotada foi fundamentada no acrônimo PECO, que consiste nas iniciais de População, Exposição, Comparador e Outcome (desfecho), tendo como pergunta norteadora: Qual é o benefício da utilização do ctDNA como ferramenta para qualificar o prognóstico de pacientes com tumores malignos sólidos do trato gastrintestinal?
Nesta revisão, foram utilizadas as bases de dados: Medical Literature Analysis and Retrievel System Online (MEDLINE), Literatura Latino-Americana e do Caribe em Ciências da Saúde (LILACS), Biblioteca Eletrônica Científica Online (SciELO), Science Direct, além do Bielefield Academic Search Engine.
Os descritores utilizados para a busca de artigos científicos neste estudo consistiram nos termos controlados do Medical Subject Headings (MeSH): “circulating tumor dna”, “liquid biopsy”, “prognosis”, “gastrointestinal cancer”, “esophageal cancer”, “gastric cancer”, “intestine cancer” e “rectal cancer”. Os seus termos alternativos também foram buscados. O operador booleano OR foi usado entre os termos controlados e seus termos alternativos, enquanto o operador AND foi usado entre os componentes do acrônimo PECO explicado anteriormente, sendo essa estratégia aplicada nos campos de busca avançada em cada base de dados. Não houve restrição de ano de publicação, idioma ou localização geográfica.
Foram inclusos neste estudo artigos do tipo caso-controle e do tipo coorte que avaliaram a eficácia da medida dos valores de ctDNA na determinação do prognóstico de pacientes com tumores malignos sólidos do trato gastrintestinal (cânceres de esôfago, estômago, intestinos e reto), sem distinção de idade ou sexo.
Foram excluídas pesquisas qualitativas, bem como outros tipos de revisão de literatura, além de artigos em que os níveis de ctDNA foram relacionados a processos metastáticos ou a neoplasias das glândulas acessórias do TGI. A seleção dos estudos foi realizada por dois pesquisadores independentes, de modo que, em caso de discordância entre estes quanto à inclusão ou exclusão de artigos, houve um terceiro revisor.
Após a busca nas bases de dados, os trabalhos recuperados foram alocados no aplicativo Rayyan, cuja finalidade é auxiliar na construção de revisões sistemáticas com metanálise8. Feita a exclusão de trabalhos duplicados, foi conduzida, inicialmente, uma leitura de títulos e resumos seguida da avaliação do texto completo. Houve cegamento na seleção dos artigos e os estudos foram selecionados pelo aplicativo supracitado. Todas estas etapas foram feitas de modo independente.
A extração dos dados, que contou com a utilização de planilha em Microsoft Excel, também foi realizada por dois pesquisadores independentes e com cegamento na extração. Os dados obtidos foram: autores, ano de publicação, periódico, país, objetivo, delineamento do estudo, local e período de coleta, tamanho da amostra e do controle, ferramentas de prognóstico e principais resultados.
A avaliação da qualidade metodológica dos artigos foi feita de acordo com o critério de Newcastle-Ottawa. Esta avaliação foi feita por dois autores de forma independente para cada um dos estudos. Para apresentação dos resultados, foi elaborada uma síntese dos dados qualitativos dos estudos que foram incluídos, por meio da construção de quadros e tabelas.
RESULTADOS
A busca inicial nas bases de dados encontrou 557 artigos, dos quais restaram 555 após a remoção de duplicatas. Outros 454 estudos foram excluídos após leitura do título e do resumo. Dos 101 artigos lidos na íntegra, oito cumpriram os critérios de elegibilidade. Além disso, cinco novos artigos foram incorporados à análise final, identificados pela análise das referências das revisões encontradas na pesquisa inicial e relacionadas ao tema, compondo uma amostra final de 13 artigos incluídos neste estudo. O processo foi ilustrado de acordo com o fluxograma proposto pelo protocolo PRISMA (Figura 1), no qual se pressupõe a descrição dos resultados do processo de busca e seleção, incluindo número de estudos identificados na busca inicial até o número de estudos incluídos na revisão9.
A maior parte dos estudos selecionados foi realizada na China (30%), seguido por Austrália (23%), Estados Unidos da América (15%) e Dinamarca (15%), sendo a maioria destes publicados nos anos de 2022, 2021 e 2019 (69%), com o mais recente em 2023 (7,6%) e o mais antigo em 2016 (7,6%). O tamanho amostral dos estudos variou de 29 a 295 participantes. A neoplasia mais abordada foi o câncer colorretal (84,6%), seguido por câncer gástrico (15%). Somente um estudo analisou o câncer esofágico (7,6%). As características gerais dos estudos estão identificadas no Quadro 110-22.
Quanto às ferramentas de prognóstico, todos os 13 artigos (100%) utilizaram pelo menos uma ferramenta adicional à avaliação do ctDNA, sendo as principais o antígeno carcinoembrionário (CEA), presente em nove estudos (69%)10-18, e a tomografia computadorizada (TC), presente em todos os 13 estudos (100%)10-22. Tais informações podem ser visualizadas no Quadro 210-22.
Em relação aos tumores gastroesofágicos, verificou-se uma taxa de recorrência significativamente maior em pacientes ctDNA-positivos em relação aos ctDNA-negativos no contexto da janela de doença residual mínima (DRM) (dentro de 16 semanas após a cirurgia de ressecção tumoral, antes do início da terapia adjuvante)19, assim como uma pior sobrevida em três anos nos pacientes com ctDNA detectável, tanto no contexto pós-cirúrgico quanto após terapia neoadjuvante. Pacientes inicialmente ctDNA-negativos que se tornaram ctDNA-positivos após terapia neoadjuvante apresentaram pior sobrevida geral20.
A mesma tendência foi observada nos casos de câncer de cólon, nos quais a positividade do ctDNA esteve relacionada a uma pior sobrevida em três anos, se comparada à negatividade do biomarcador15,21. Li et al.21 propuseram uma nova forma de estratificação de risco em pacientes com neoplasias de cólon com base na inclusão do ctDNA aos demais fatores utilizados previamente, encontrando uma eficaz correlação dos níveis de risco com a recidiva tumoral.
Adicionalmente, o CEA esteve menos presente do que o ctDNA no momento da recidiva em pacientes com tumores de cólon em estádio II, com intervalo expressivamente maior entre a elevação do ctDNA e a evidência radiológica de recidiva tumoral em comparação ao tempo de elevação do CEA14.
Nos estudos que avaliaram o câncer colorretal, encontrou-se uma positividade mais elevada do ctDNA consistentemente nos tumores em estádios II e III em comparação àqueles em estádio I. Ainda assim, de modo geral, níveis detectáveis de ctDNA nestes pacientes estiveram associados a piores taxas de recorrência em pelo menos dois anos10,13. Complementarmente, encontrou-se um baixo risco de recidiva tumoral para pacientes ctDNA-negativos, independentemente de terem passado por terapia adjuvante ou não13. Destaca-se também, nesse contexto, a possível vantagem da utilização do ctDNA na predição de recidivas antes de sua comprovação radiológica em comparação ao CEA13,17.
No que se refere aos cânceres retais, a detecção do ctDNA também foi associada à pior sobrevida geral em três anos, tanto no cenário pós-operatório como antes mesmo do início da terapia neoadjuvante16,22. Ademais, a taxa de ressecção R0 (ressecção macroscopicamente completa com margens histologicamente negativas) foi significativamente maior entre pacientes com ctDNA pré-operatório indetectável em comparação àqueles ctDNA-positivos12.
A avaliação da qualidade metodológica segundo o Newcastle-Ottawa Quality Assessment Scale evidenciou que a média da pontuação dos estudos foi de 8,69 (desvio-padrão: ±0,87). Entre os estudos, 11 (84%) obtiveram pontuação máxima, um estudo (7,6%) somou oito pontos, e outro estudo (7,6%) obteve seis pontos, conforme pode ser verificado no Quadro 3.
DISCUSSÃO
Diante do desafio de determinar com acurácia o prognóstico do paciente oncológico, novas tecnologias têm surgido na tentativa de guiar o tratamento das neoplasias malignas de maneira mais eficaz. Entre elas, a biópsia líquida tem ganhado destaque nos anos recentes, representada predominantemente pelo ctDNA. Embora sua aplicabilidade na prática clínica ainda seja relativamente restrita – em virtude de seu alto custo e baixa disponibilidade, sobretudo em países em desenvolvimento –, diversos estudos discorrem sobre seu caráter promissor na avaliação de prognóstico e estratificação de risco, com possibilidade de identificar pacientes com DRM e guiar a necessidade de terapia adjuvante, bem como verificar sua efetividade23,24.
Os artigos analisados nesta revisão verificaram uma correlação positiva entre a detecção do ctDNA e prognósticos desfavoráveis em pacientes com tumores malignos sólidos do trato gastrintestinal, em comparação àqueles ctDNA-negativos. No caso dos tumores gastroesofágicos, não somente a recorrência tumoral foi maior em pacientes ctDNA-positivos, como também houve pior sobrevida em três anos. Nos tumores colorretais, a mesma tendência foi observada: houve pior sobrevida em três anos e piores taxas de recorrência, além de um maior espaço entre a elevação dos níveis de ctDNA e a comprovação radiológica de recidiva em comparação à verificação dos níveis de CEA para o mesmo propósito.
Jiang et al.25, ao conduzirem uma revisão da literatura sobre o prognóstico do câncer esofágico, constataram que o ctDNA detectável após tratamento, em qualquer time point, correlacionou-se com pior prognóstico no geral, o que corrobora os resultados obtidos nesse estudo. Similarmente, Gao et al.26, ao realizar uma revisão sistemática com metanálise que discorreu sobre o câncer gástrico, destacaram que a detecção de ctDNA também esteve associada a piores taxas de sobrevida livre de recorrência e de sobrevida geral. Embora somente quatro artigos pudessem ter sido avaliados quanto a este último parâmetro, o trabalho contou com metodologia semelhante ao do presente estudo, o que reforça os achados descritos.
Quanto ao câncer colorretal, outra revisão sistemática da literatura afirma que a positividade do ctDNA correlacionou-se da mesma maneira com as taxas previamente citadas. Destaca-se que o estudo se debruçou sobre algumas das mutações genéticas e epigenéticas encontradas no ctDNA e como estas poderiam se relacionar com o prognóstico do paciente, relatando que em seus resultados houve um discreto nível de heterogeneidade dessa avaliação27.
A análise do ctDNA por next generation sequencing (NGS) realizada com auxílio de painel genético, por exemplo, é capaz de identificar variações conhecidas nos genes de alguns tumores28. Ao fazer uso da detecção de algumas dessas alterações, a biópsia líquida permite avaliar de modo qualitativo o ctDNA, correlacionando alterações genéticas específicas a prognósticos mais ou menos favoráveis29.
Chen et al.30, em sua revisão sistemática com metanálise, observaram que a positividade do ctDNA apresentou-se como um importante indicador de recidiva dos tumores colorretais, um padrão observado em todos os estudos analisados, que não se alterou com tempo e local do estudo e que apresenta maior eficácia na avaliação de prognóstico dos tumores em estádios I a III. Ressalta-se, nesse caso, o fato de que cinco dos artigos incluídos nessa metanálise também foram contemplados no escopo do presente estudo, reforçando assim a convergência dos resultados obtidos.
A monitorização durante tratamentos oncológicos pode revelar uma redução importante dos níveis de ctDNA após intervenção inicial, evidenciando uma resposta clínica adequada31. Em contrapartida, a subsequente elevação do ctDNA durante o acompanhamento longitudinal configura-se como possível indício de DRM e consequente insucesso terapêutico32.
Além disso, um trabalho publicado em 2019, cujos objetivos incluem a avaliação do ctDNA como ferramenta para avaliação de prognóstico em pacientes com tumores retais, encontrou similar correlação entre a persistência do ctDNA após tratamento e a recorrência tumoral no período de follow-up na grande maioria dos artigos inclusos em sua revisão33.
No que diz respeito às suas limitações, o presente estudo lida com problemas de natureza da própria avaliação do ctDNA. Atualmente, ainda não há um consenso sobre qual a maneira mais eficaz de obtenção desse biomarcador, de modo que se encontra, na literatura, uma grande heterogeneidade de tecnologias, como o NGS, o digital PCR (dPCR) e o cancer personalized profiling by deep sequencing (CAPP-Seq), todos capazes de realizar essa análise23. Igualmente, não há regularidade exata quanto ao tempo de coleta do ctDNA em relação à terapia neoadjuvante, cirurgia e terapia adjuvante, inconsistências as quais são naturalmente encontradas ao longo dos trabalhos abordados.
CONCLUSÃO
O ctDNA, um dos principais expoentes da biópsia líquida, apresenta indícios de sucesso na avaliação do prognóstico de pacientes com tumores malignos sólidos do trato gastrintestinal. Os resultados encontrados nesse estudo sugerem que existem, na literatura recente, evidências para suportar sua aplicabilidade na prática clínica no cenário específico descrito, sobretudo de maneira complementar às ferramentas de avaliação de prognóstico mais comumente utilizadas.
Futuramente, novos estudos podem buscar uma maior uniformização na forma como se coleta e se avalia o ctDNA, em vista das diferentes possibilidades que atualmente estão disponíveis. Ademais, a realização de estudos multicêntricos, capazes de testar a aplicabilidade do ctDNA em regiões onde a prática tem sido menos explorada – como América Latina – podem fornecer mais subsídio para a aplicação dessa ferramenta na área clínica, ao verificar a provável persistência do seu potencial em cenários nos quais as características populacionais sejam distintas.
CONTRIBUIÇÕES
Gabriel dos Santos Martins de Abreu e Mariana Albuquerque de Brito contribuíram substancialmente na concepção e no planejamento do estudo; na obtenção, análise e interpretação dos dados; e na redação. Ana Paula de Souza Ramos contribuiu substancialmente na obtenção, análise e interpretação dos dados; na redação e na revisão crítica. Todos os autores aprovaram a versão final a ser publicada.
DECLARAÇÃO DE CONFLITOS DE INTERESSES
Nada a declarar.
FONTES DE FINANCIAMENTO
REFERÊNCIAS
1. American Cancer Society. Global cancer facts & figures. 5 ed. Nova Yorke: ACS; 2024.
2. Souza T, Migowski A, Ribeiro C, et al. ABC do câncer: abordagens básicas para o controle do câncer. 6 ed rev atual. Rio de Janeiro: INCA; 2020.
3. Vaidyanathan R, Soon RH, Zhang P, et al. Cancer diagnosis: from tumor to liquid biopsy and beyond. Lab Chip. 2019;19:11-34. doi: https://doi.org/10.1039/c8lc00684a
4. Pessoa LS, Heringer M, Ferrer VP. ctDNA as a cancer biomarker: a broad overview. Crit Rev Oncol Hematol. 2020;155:103109. doi: https://doi.org/10.1016/j.critrevonc.2020.103109
5. Leso H, Moraes J, Amorim I, et al. Epidemiologia do câncer do trato gastrointestinal em itumbiara, Goiás, entre 1999 e 2019. RSD. 2022;11(15). doi: https://doi.org/10.33448/rsd-v11i15.37540
6. Shamseer L, Moher D, Clarke M, et al. Preferred reporting items for systematic review and meta-analysis protocols (PRISMA-P) 2015: elaboration and explanation. BMJ. 2015;350:g7647. doi: https://doi.org/10.1136/bmj.g7647
7. University of York. Centre for Reviews and Dissemination. New York: University of York; 2019. PROSPERO - International prospective register of systematic reviews. 2023. [acesso 2023 ago 31]. Disponível em: https://www.crd.york.ac.uk/PROSPERO/
8. Ouzzani M, Hammady H, Fedorowicz Z, et al. Rayyan-a web and mobile app for systematic reviews. Syst Rev. 2016;5(1):210. doi: https://doi.org/10.1186/s13643-016-0384-4
9. Page MJ, McKenzie JE, Bossuyt PM, et al. A declaração PRISMA 2020: diretriz atualizada para relatar revisões sistemáticas. Epidemiol Serv Saúde. 2022;31(2):e2022107. doi: http://dx.doi.org/10.1590/s1679-49742022000200033
10. Chen G, Peng J, Xiao Q, et al. Postoperative circulating tumor DNA as markers of recurrence risk in stages II to III colorectal cancer. J Hematol Oncol. 2021;14(1):80. doi: https://doi.org/10.1186/s13045-021-01089-z
11. Henriksen TV, Tarazona N, Frydendahl A, et al. Circulating tumor DNA in stage III colorectal cancer, beyond minimal residual disease detection, toward assessment of adjuvant therapy efficacy and clinical behavior of recurrences. Clin Cancer Res. 2022;28(3):507-17. doi: https://doi.org/10.1158/1078-0432.ccr-21-2404
12. McDuff SGR, Hardiman KM, Ulintz PJ, et al. Circulating tumor DNA predicts pathologic and clinical outcomes following neoadjuvant chemoradiation and surgery for patients with locally advanced rectal cancer. JCO Precis Oncol. 2021;5:PO.20.00220. doi: https://doi.org/10.1200/po.20.00220
13. Reinert T, Henriksen TV, Christensen E, et al. Analysis of plasma cell-free DNA by ultradeep sequencing in patients with stages I to III colorectal cancer. JAMA Oncol. 2019;5(8):1124-31. doi: https://doi.org/10.1001/jamaoncol.2019.0528
14. Tie J, Wang Y, Tomasetti C, et al. Circulating tumor DNA analysis detects minimal residual disease and predicts recurrence in patients with stage II colon cancer. Sci Transl Med. 2016;8(346):346ra92. doi: https://doi.org/10.1126/scitranslmed.aaf6219
15. Tie J, Cohen JD, Wang Y, et al. Circulating tumor DNA analyses as markers of recurrence risk and benefit of adjuvant therapy for stage III colon cancer. JAMA Oncol. 2019;5(12):1710-7. doi: https://doi.org/10.1001/jamaoncol.2019.3616
16. Tie J, Cohen JD, Wang Y, et al. Serial circulating tumour DNA analysis during multimodality treatment of locally advanced rectal cancer: a prospective biomarker study. Gut. 2019;68(4):663-71. doi: https://doi.org/10.1136/gutjnl-2017-315852
17. Wang Y, Li L, Cohen JD, et al. Prognostic potential of circulating tumor DNA measurement in postoperative surveillance of nonmetastatic colorectal cancer. JAMA Oncol. 2019;5(8):1118-23. doi: https://doi.org/10.1001/jamaoncol.2019.0512
18. Zhou J, Wang C, Lin G, et al. Serial circulating tumor DNA in predicting and monitoring the effect of neoadjuvant chemoradiotherapy in patients with rectal cancer: a prospective multicenter study. Clin Cancer Res. 2021;27(1):301-10. doi: https://doi.org/10.1158/1078-0432.ccr-20-2299
19. Huffman BM, Aushev VN, Budde GL, et al. Analysis of circulating tumor DNA to predict risk of recurrence in patients with esophageal and gastric cancers. JCO Precis Oncol. 2022;(6):e2200420. doi: https://doi.org/10.1200/po.22.00420
20. Zhang M, Yang H, Fu T, et al. Liquid biopsy: circulating tumor DNA monitors neoadjuvant chemotherapy response and prognosis in stage II/III gastric cancer. Mol Oncol. 2023;17(9):1930-42. doi: https://doi.org/10.1002/1878-0261.13481
21. Li Y, Mo S, Zhang L, et al. Postoperative circulating tumor DNA combined with consensus molecular subtypes can better predict outcomes in stage III colon cancers: a prospective cohort study. Eur J Cancer. 2022;169:198-209. doi: https://doi.org/10.1016/j.ejca.2022.04.010
22. Pazdirek F, Minarik M, Benesova L, et al. Monitoring of early changes of circulating tumor dna in the plasma of rectal cancer patients receiving neoadjuvant concomitant chemoradiotherapy: evaluation for prognosis and prediction of therapeutic response. Front Oncol. 2020;10:1028. doi: https://doi.org/10.3389/fonc.2020.01028
23. Peng Y, Mei W, Ma K, et al. Circulating tumor DNA and minimal residual disease (MRD) in solid tumors: current horizons and future perspectives. Front Oncol. 2021;11:763790. doi: https://doi.org/10.3389/fonc.2021.763790
24. Ueberroth BE, Jones JC, Bekaii-Saab TS. Circulating tumor DNA (ctDNA) to evaluate minimal residual disease (MRD), treatment response, and posttreatment prognosis in pancreatic adenocarcinoma. Pancreatology. 2022;22(6):741-8. doi: https://doi.org/10.1016/j.pan.2022.06.009
25. Jiang M, Zhou H, Jiang S, et al. A review of circulating tumor DNA in the diagnosis and monitoring of esophageal cancer. Med Sci Monit. 2022;28:e934106. doi: https://doi.org/10.12659/msm.934106
26. Gao Y, Zhang K, Xi H, et al. Diagnostic and prognostic value of circulating tumor DNA in gastric cancer: a meta-analysis. Oncotarget. 2016;8(4):6330-40. doi: https://doi.org/10.18632/oncotarget.14064
27. Fan G, Zhang K, Yang X, et al. Prognostic value of circulating tumor DNA in patients with colon cancer: systematic review. PLoS One. 2017;12(2):e0171991. doi: https://doi.org/10.1371/journal.pone.0171991
28. Cimmino F, Lasorsa VA, Vetrella S, et al. A targeted gene panel for circulating tumor DNA sequencing in neuroblastoma. Front Oncol. 2020;10:596191. doi: https://doi.org/10.3389/fonc.2020.596191
29. Lim Y, Kim S, Kang JK, et al. Circulating tumor DNA sequencing in colorectal cancer patients treated with first-line chemotherapy with anti-EGFR. Sci Rep. 2021;11:16333. doi: https://doi.org/10.1038/s41598-021-95345-4
30. Chen Y, Mo S, Wu M, et al. Circulating tumor DNA as a prognostic indicator of colorectal cancer recurrence - a systematic review and meta-analysis. Int J Colorectal Dis. 2022;37(5):1021-7. doi: https://doi.org/10.1007/s00384-022-04144-4
31. Cheng ML, Lau CJ, Milan MSD, et al. Plasma ctDNA response is an early marker of treatment effect in advanced NSCLC. JCO Precis Oncol. 2021;5:PO.20.00419. doi: https://doi.org/10.1200/po.20.00419
32. Sharbatoghli M, Vafaei S, Aboulkheyr ESH, et al. Prediction of the treatment response in ovarian cancer: a ctDNA approach. J Ovarian Res. 2020;13(1):124. doi: https://doi.org/10.1186/s13048-020-00729-1
33. Massihnia D, Pizzutilo EG, Amatu A, et al. Liquid biopsy for rectal cancer: a systematic review. Cancer Treat Rev. 2019;79:101893. doi: https://doi.org/10.1016/j.ctrv.2019.101893
Recebido em 20/8/2024
Aprovado em 3/12/2024
Editor-associado: Fernando Lopes Tavares de Lima. Orcid iD: https://orcid.org/0000-0002-8618-7608
Editora-científica: Anke Bergmann. Orcid iD: https://orcid.org/0000-0002-1972-8777
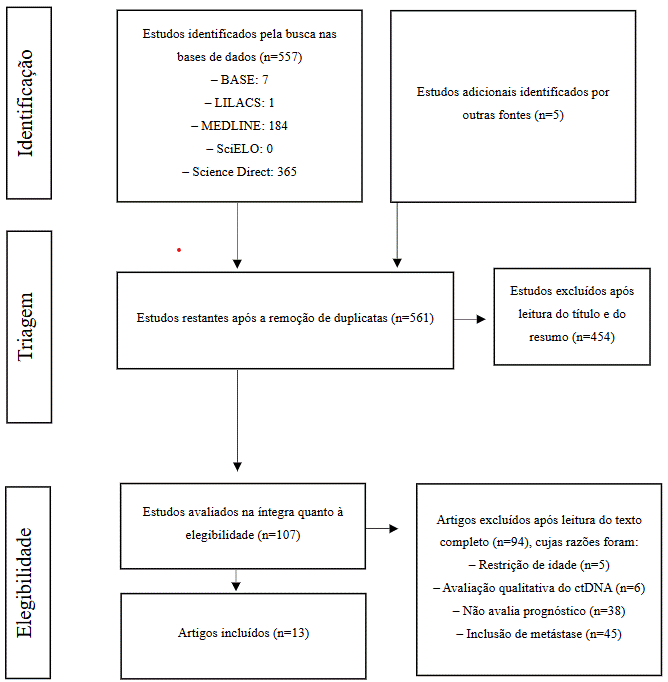
Figura 1. Fluxograma PRISMA
Legenda: ctDNA = DNA tumoral circulante.
Quadro 1. Características gerais dos estudos inclusos
|
Autor |
Ano |
Período de coleta |
País |
Delineamento do estudo |
Tamanho amostral |
Tipo de tumor |
|
Chen et al.10 |
2021 |
Set 2017 – Mar 2020 |
China |
Coorte |
276 |
Colorretal |
|
Henriksen et al.11 |
2022 |
Jul 2014 – Fev 2019 |
Dinamarca |
Coorte |
168 |
Colorretal |
|
McDuff et al.12 |
2021 |
Jan 2014 – Fev 2018 |
Estados Unidos |
Coorte |
29 |
Colorretal |
|
Reinert et al.13 |
2019 |
Mai 2014 – Jan 2017 |
Dinamarca |
Coorte |
130 |
Colorretal |
|
Tie et al.14 |
2016 |
Jul 2011 – Set 2014 |
Austrália |
Coorte |
250 |
Colorretal |
|
Tie et al.15 |
2019 |
Nov 2014 – Mai 2017 |
Austrália |
Coorte |
100 |
Colorretal |
|
Tie et al.16 |
2019 |
Abr 2012 – Dez 2015 |
Austrália |
Coorte |
200 |
Colorretal |
|
Wang et al.17 |
2019 |
Fev 2007 – Mai 2013 |
Suécia |
Coorte |
63 |
Colorretal |
|
Zhou et al.18 |
2021 |
Ag 2017 – Fev 2019 |
China |
Coorte |
106 |
Colorretal |
|
Huffman et al.19 |
2022 |
Set 2019 – Fev 2022 |
EUA |
Coorte |
295 |
Esofágico e gástrico |
|
Zhang et al.20 |
2023 |
Nov 2017 – Jan 2020 |
China |
Coorte |
79 |
Gástrico |
|
Li et al.21 |
2022 |
Ago 2018 – Dez 2019 |
China |
Coorte |
165 |
Colorretal |
|
Pazdirek et al.22 |
2020 |
2013 – 2017 |
Tchéquia |
Coorte |
36 |
Colorretal |
Quadro 2. Ferramentas de prognóstico e principais resultados dos estudos inclusos
|
Autor |
Ano |
|
|
Ferramentas de prognóstico |
Principais resultados |
|
Chen et al.10 |
2021 |
|
|
ctDNA, CEA, TC |
No pré-operatório, o ctDNA foi detectável em 64,2% dos pacientes; no pós-operatório, a negatividade do ctDNA esteve associada a uma taxa de sobrevida livre de recorrência em dois anos de 89,4%, em contraste aos 39,9% dos pacientes ctDNA-positivos. As amostras de ctDNA foram coletadas no pré-operatório, pós-operatório e seis meses após a cirurgia de modo seriado a cada três meses até o 24º mês ou retirada ou óbito do paciente, sendo analisadas por next generation sequencing |
|
Henriksen et al.11 |
2022 |
|
|
ctDNA, CEA, TC |
Houve uma taxa de recorrência de 80% nos pacientes ctDNA-positivos no pós-operatório e, nesse contexto, o ctDNA superou idade ≥ 70 anos, T4, grau de diferenciação tumoral e CEA como preditor de sobrevida livre de recorrência; após terapia adjuvante, 100% dos pacientes que não negativaram ctDNA no período de follow-up recidivaram. As amostras de ctDNA foram coletadas no diagnóstico, no pós-operatório, durante terapia adjuvante e após seu término e no acompanhamento de rotina, a cada três meses por até três anos, sendo analisadas por next generation sequencing |
|
McDuff et al.12 |
2021 |
|
|
ctDNA, CEA, TC |
A taxa de ressecção R0 (ressecção macroscopicamente completa com margens histologicamente negativas) foi significativamente maior entre pacientes com ctDNA pré-operatório indetectável (88%) em comparação àqueles ctDNA-positivos (44%). As amostras de ctDNA foram coletadas, de modo único, no pré-operatório e no pós-operatório, sendo analisadas por next generation sequencing |
|
Reinert et al.13 |
2019 |
|
|
ctDNA, CEA, TC |
A detecção do ctDNA nos tumores em estádios II e III foi significativamente maior em relação àqueles em estádio I; o tempo médio da detecção de níveis elevados de ctDNA nos pacientes que completaram o tratamento definitivo até a evidência de recidiva por meio da TC foi de 8,7 meses. As amostras de ctDNA foram coletadas antes e após a intervenção cirúrgica, analisadas por next generation sequencing e seriadas a cada três meses até óbito, retirada do paciente ou até o 36º mês |
|
Tie et al.14 |
2016 |
|
|
ctDNA, CEA, TC |
Recorrência radiológica foi detectada em 78,6% de pacientes ctDNA-positivos não tratados com terapia adjuvante; no período de follow-up, o ctDNA foi mais frequentemente positivo do que o CEA no momento da recidiva, além de apresentar elevação em seus níveis em um tempo significativamente maior do que o CEA até a evidência radiológica de recorrência tumoral. As amostras de ctDNA foram coletadas após intervenção cirúrgica, analisadas por safe-sequencing system e seriadas a cada três meses por até dois anos |
|
Tie et al.15 |
2019 |
|
|
ctDNA, CEA, TC |
A sobrevida livre de recorrência em três anos foi de 33% para pacientes ctDNA-positivos no pós-operatório e de 87% nos ctDNA-negativos; padrão semelhante foi encontrado também após terapia neoadjuvante (50% e 85%, respectivamente). As amostras de ctDNA foram coletadas, de modo único, antes do início do pré-tratamento (radioquimioterapia), após seu final e 4-10 semanas após intervenção cirúrgica, sendo analisadas por safe-sequencing system |
|
Tie et al.16 |
2019 |
|
|
ctDNA, CEA, TC |
Após cirurgia, a sobrevida em três anos nos pacientes ctDNA-positivos foi de 47%; o status pós-cirúrgico do ctDNA possuiu a mais forte associação independente com o intervalo livre de recorrências. As amostras de ctDNA foram coletadas de modo único após intervenção cirúrgica e ao final do tratamento, sendo analisadas por safe-sequencing system |
|
Wang et al.17 |
2019 |
|
|
ctDNA, CEA, TC |
No pós-operatório, houve positividade do ctDNA entre 2 a 31 meses antes da comprovação radiológica de recidiva tumoral; 100% dos pacientes que recidivaram apresentaram ctDNA detectável no período de follow-up. As amostras de ctDNA foram coletadas um mês após intervenção cirúrgica, seriadas a cada três a seis meses e analisadas por safe-sequencing system |
|
Zhou et al.18 |
2021 |
|
|
ctDNA, CEA, CA19-9, TC |
A detecção de ctDNA na linha de base se correlacionou positivamente com a ocorrência de metástases a distância em um período de tempo menor do que aqueles pacientes cujo ctDNA foi indetectável. Essa correlação foi positiva em todos os momentos de dosagem. As amostras de ctDNA foram coletadas de modo seriado na linha de base, durante a radioquimioterapia neoadjuvante, no pré-operatório e no pós-operatório, sendo analisadas por next generation sequencing |
|
Huffman et al.19 |
2022 |
|
|
ctDNA, TC |
O ctDNA esteve presente em cerca de um quinto dos pacientes no contexto pós-operatório, associando-se a uma taxa de recorrência de 81,2%; em todos os cenários analisados, houve semelhança dos resultados nos diversos subtipos tumorais. As amostras de ctDNA foram coletadas no pré-operatório, pós-operatório e em série durante acompanhamento clínico de rotina a critério médico, sendo analisadas por next generation sequencing |
|
Zhang et al.20 |
2023 |
|
|
ctDNA, TC |
A estimativa de sobrevida em três anos para os pacientes ctDNA-negativos e ctDNA-positivos foi de 73% e 34%, respectivamente, após terapia neoadjuvante e de 68% e 38%, respectivamente, após cirurgia. As amostras de ctDNA foram coletadas, de modo único, antes da quimioterapia neoadjuvante, após quimioterapia neoadjuvante e após cirurgia, sendo analisadas por next generation sequencing |
|
Li et al.21 |
2022 |
|
|
ctDNA, TC |
A sobrevida livre de recorrências nos pacientes ctDNA-positivos pós-quimioterapia adjuvante foi de 45,5%, em contraste aos 72,7% dos ctDNA-negativos. As amostras de ctDNA foram coletadas antes do início da quimioterapia e após seu final de modo não seriado, sendo analisadas por next generation sequencing |
|
Pazdirek et al.22 |
2020 |
|
|
ctDNA, TC |
A taxa de sobrevida global em pacientes cujo ctDNA foi negativo foi de 91,2%. A taxa do subgrupo de pacientes cujo ctDNA foi positivo correndeu a 71,4%. As amostras de ctDNA foram coletadas de modo seriado antes da terapia e ao final da primeira semana. As tecnologias utilizadas para análise foram eletroforese capilar desnaturante e ensaio BEAMing |
Legendas: ctDNA = DNA tumoral circulante, CEA = antígeno carcinoembrionário, CA19-9 = antígeno carboidrato 19-9, TC = tomografia computadorizada.
Quadro 3. Avaliação de qualidade metodológica por Newcaslte-Ottawa Quality Assessment Scale
|
Autor |
Ano |
Seleção |
Comparabilidade |
Desfecho |
Total |
|
Chen et al.10 |
2021 |
++++ |
++ |
+++ |
9/9 |
|
Henriksen et al.11 |
2022 |
++++ |
++ |
+++ |
9/9 |
|
McDuff et al.12 |
2021 |
++ |
++ |
++ |
6/9 |
|
Reinert et al.13 |
2019 |
++++ |
++ |
+++ |
9/9 |
|
Tie et al.14 |
2016 |
++++ |
++ |
+++ |
9/9 |
|
Tie et al.15 |
2019 |
++++ |
++ |
+++ |
9/9 |
|
Tie et al.16 |
2019 |
++++ |
++ |
+++ |
9/9 |
|
Wang et al.17 |
2019 |
++++ |
++ |
+++ |
9/9 |
|
Zhou et al.18 |
2021 |
++++ |
++ |
+++ |
9/9 |
|
Huffman et al.19 |
2022 |
++++ |
++ |
+++ |
9/9 |
|
Zhang et al.20 |
2023 |
++++ |
++ |
++ |
8/9 |
|
Li et al.21 |
2022 |
++++ |
++ |
+++ |
9/9 |
|
Pazdirek et al.22 |
2020 |
++++ |
++ |
+++ |
9/9 |
![]()
Este é um artigo publicado em acesso aberto (Open Access) sob a licença Creative Commons Attribution, que permite uso, distribuição e reprodução em qualquer meio, sem restrições, desde que o trabalho original seja corretamente citado.